En regresión múltiple, si alguna de las variables explicativas no aporta información adicional relevante sobre la respuesta puede aparecer el problema de la colinealidad (o concurvidad, si se consideran efectos no lineales).
En regresión lineal múltiple se supone que ninguna de las variables explicativas es combinación lineal de las demás. Si una de las variables explicativas (variables independientes) es combinación lineal de las otras, no se pueden determinar los parámetros de forma única (sistema singular). Sin llegar a esta situación extrema, cuando algunas variables explicativas estén altamente correlacionadas entre sí, tendremos una situación de alta colinealidad. En este caso las estimaciones de los parámetros pueden verse seriamente afectadas:
Tendrán varianzas muy altas (serán poco eficientes).
Habrá mucha dependencia entre ellas (al modificar ligeramente el modelo, añadiendo o eliminando una variable o una observación, se producirán grandes cambios en las estimaciones de los efectos).
Consideraremos un ejemplo de regresión lineal bidimensional con datos simulados en el que las dos variables explicativas están altamente correlacionadas:
set.seed(1)
n <- 50
rand.gen <- runif # rnorm
x1 <- rand.gen(n)
rho <- sqrt(0.99) # coeficiente de correlación
x2 <- rho*x1 + sqrt(1 - rho^2)*rand.gen(n)
fit.x2 <- lm(x2 ~ x1)
# plot(x1, x2)
# summary(fit.x2)
# Rejilla x-y para predicciones:
x1.range <- range(x1)
x1.grid <- seq(x1.range[1], x1.range[2], length.out = 30)
x2.range <- range(x2)
x2.grid <- seq(x2.range[1], x2.range[2], length.out = 30)
xy <- expand.grid(x1 = x1.grid, x2 = x2.grid)
# Modelo teórico:
model.teor <- function(x1, x2) x1
# model.teor <- function(x1, x2) x1 - 0.5*x2
y.grid <- matrix(mapply(model.teor, xy$x1, xy$x2), nrow = length(x1.grid))
y.mean <- mapply(model.teor, x1, x2)Tendencia teórica y valores de las variables explicativas:
library(plot3D)
ylim <- c(-2, 3) # range(y, y.pred)
scatter3D(z = y.mean, x = x1, y = x2, pch = 16, cex = 1, clim = ylim, zlim = ylim,
theta = -40, phi = 20, ticktype = "detailed",
main = "Modelo teórico y valores de las variables explicativas",
xlab = "x1", ylab = "x2", zlab = "y", sub = sprintf("R2(x1,x2) = %.2f", summary(fit.x2)$r.squared),
surf = list(x = x1.grid, y = x2.grid, z = y.grid, facets = NA))
scatter3D(z = rep(ylim[1], n), x = x1, y = x2, add = TRUE, colkey = FALSE,
pch = 16, cex = 1, col = "black")
x2.pred <- predict(fit.x2, newdata = data.frame(x1 = x1.range))
lines3D(z = rep(ylim[1], 2), x = x1.range, y = x2.pred, add = TRUE, colkey = FALSE, col = "black") 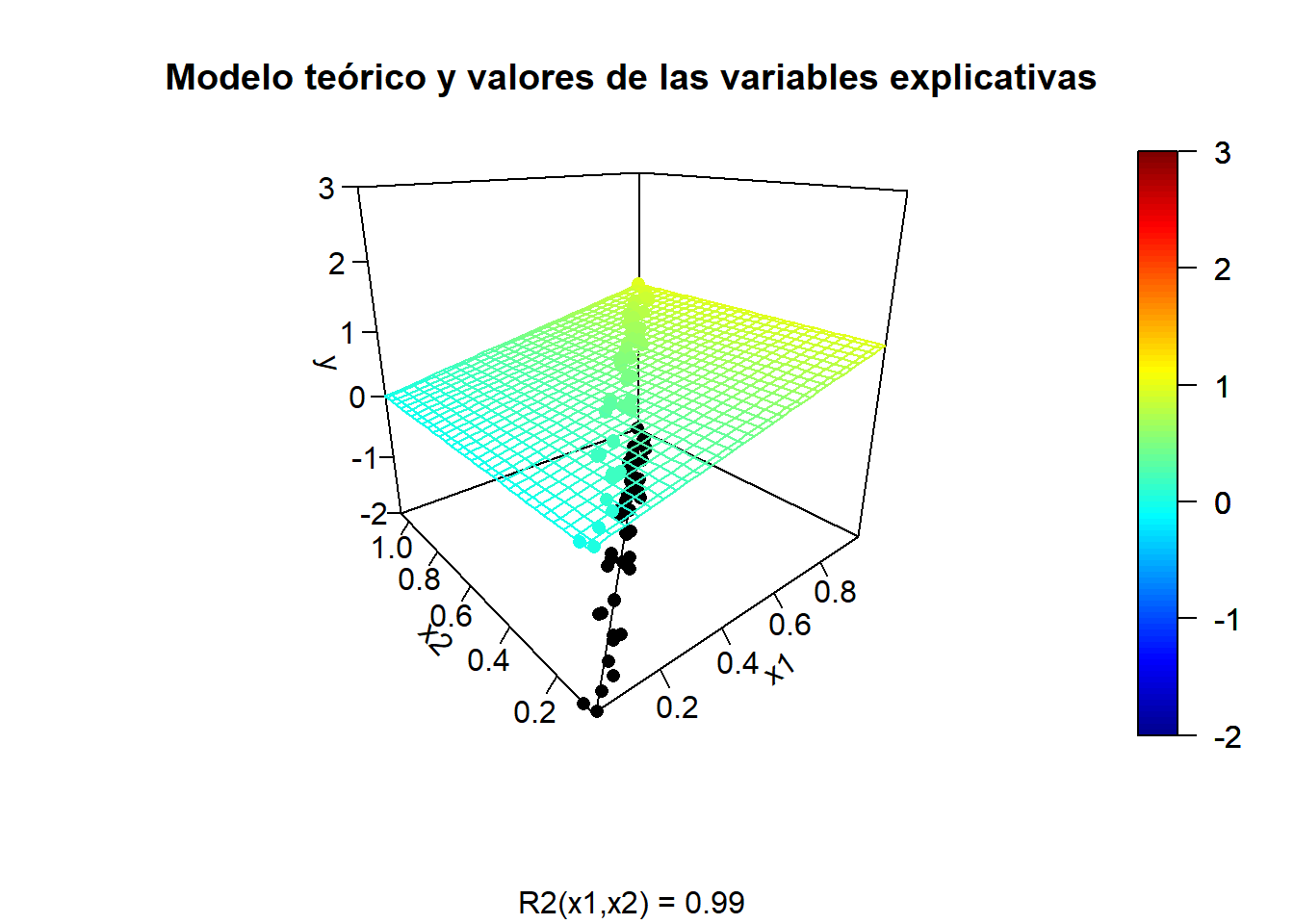
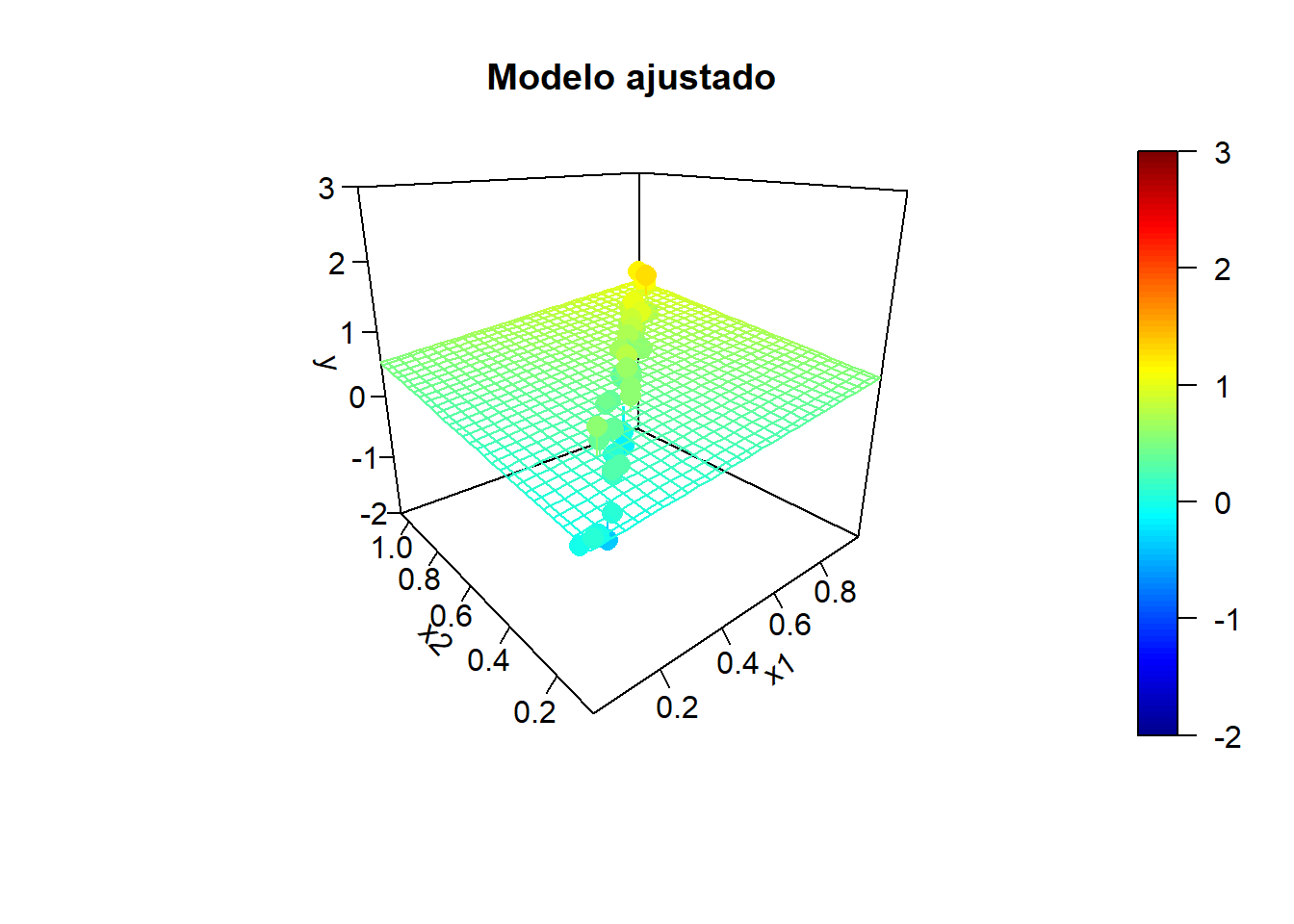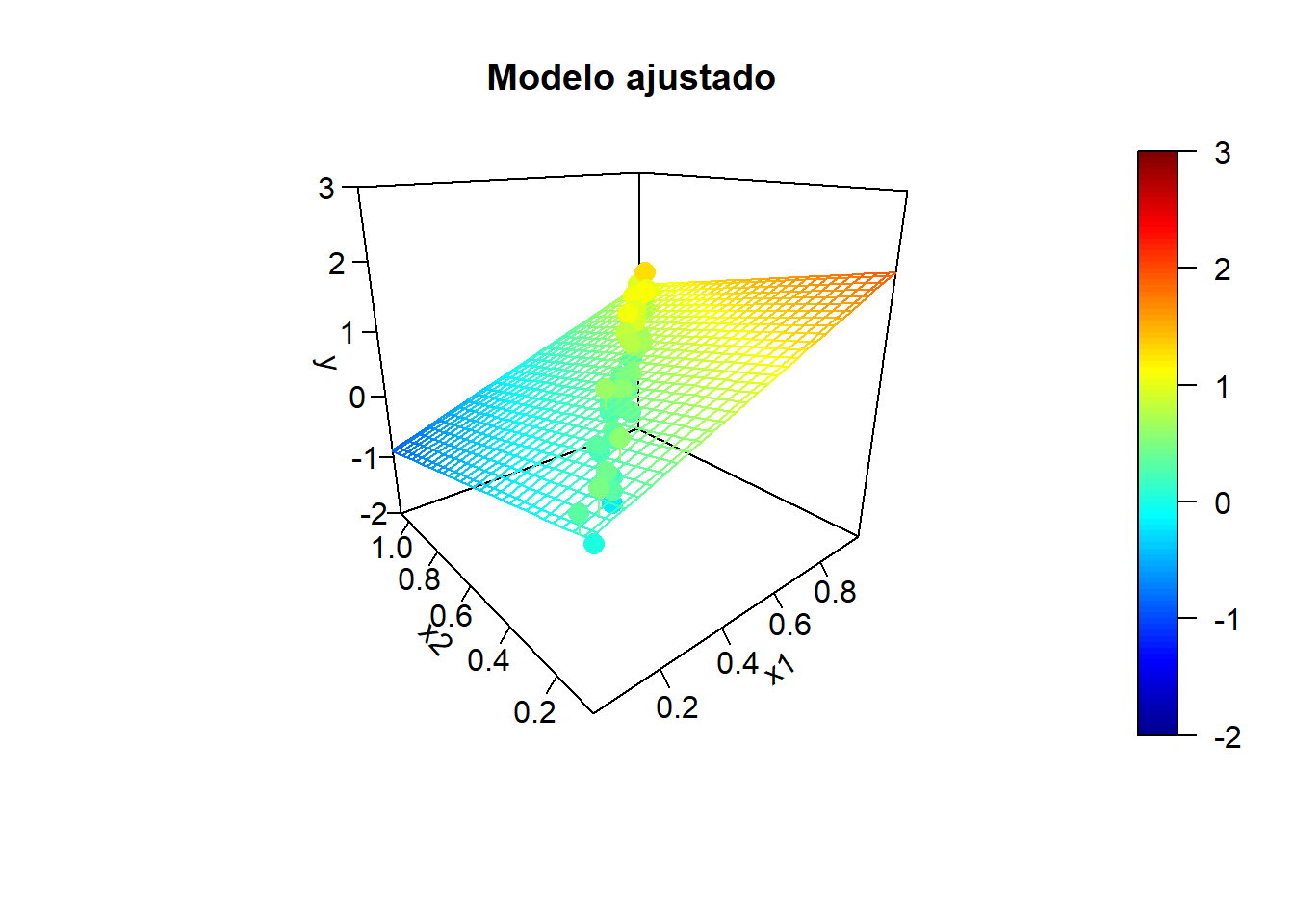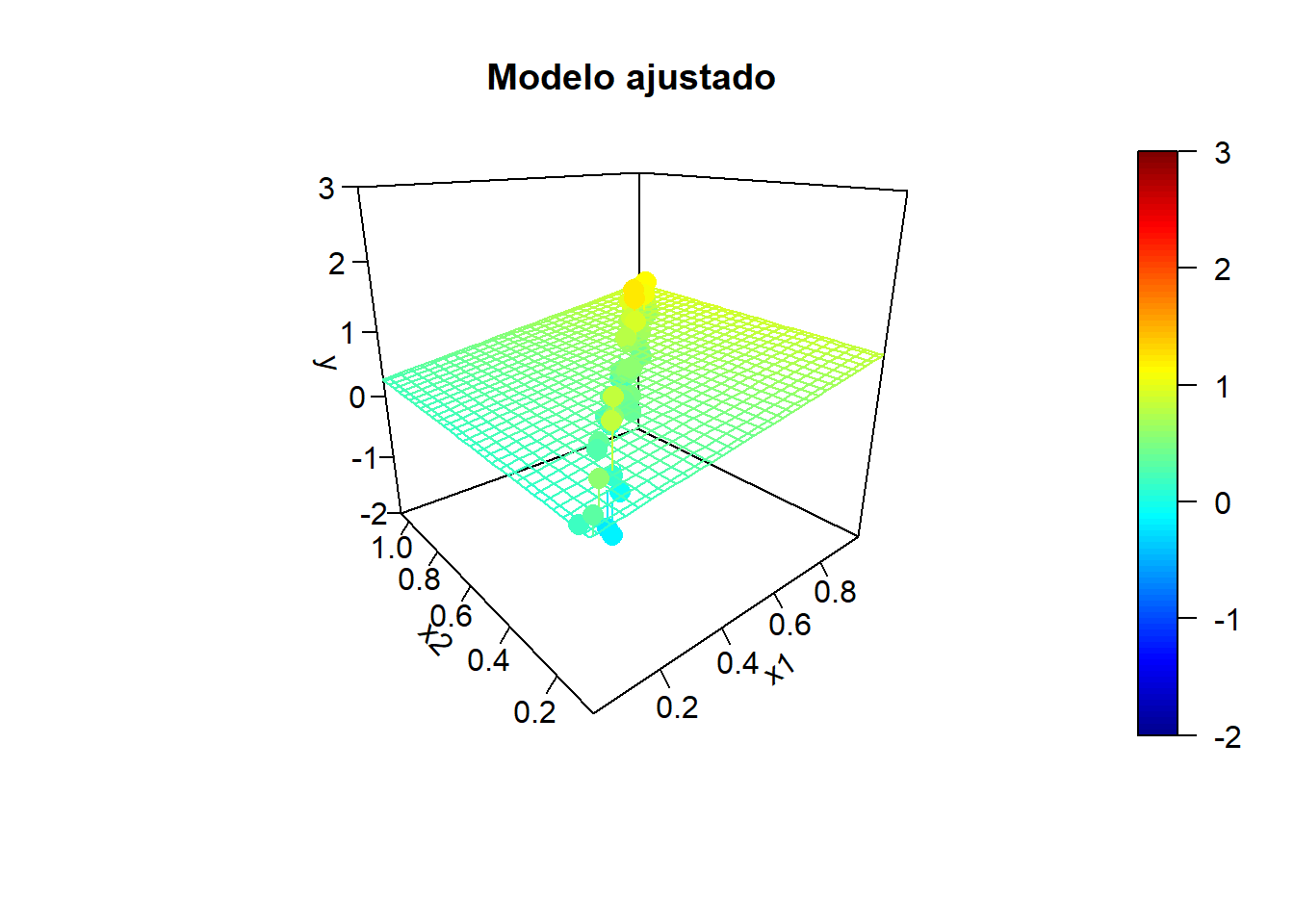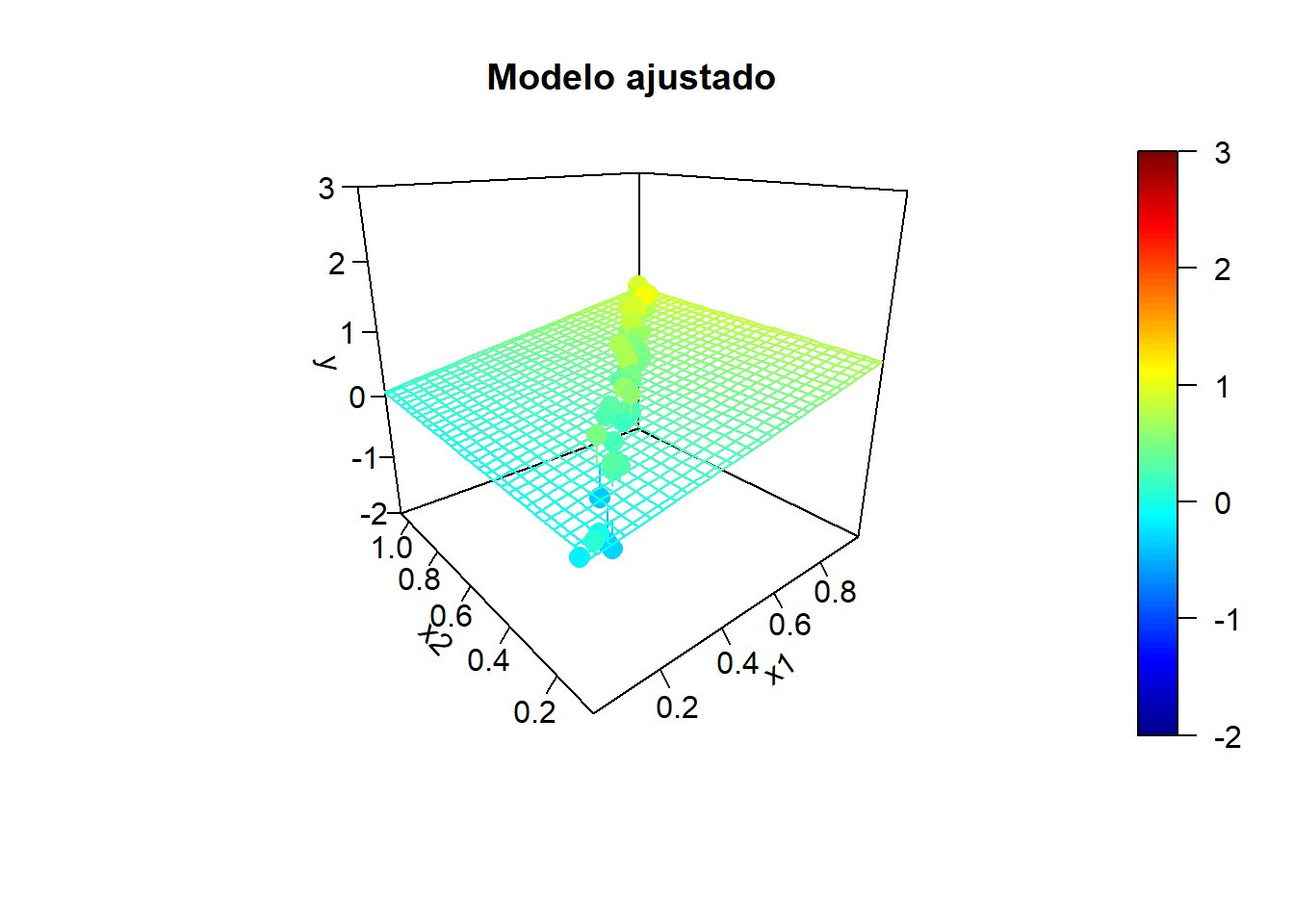
Simulación de la respuesta:
sd.err <- 0.25
nsim <- 10
for (isim in 1:nsim) {
set.seed(isim)
y <- y.mean + rnorm(n, 0, sd.err)
# Ajuste lineal y superficie de predicción
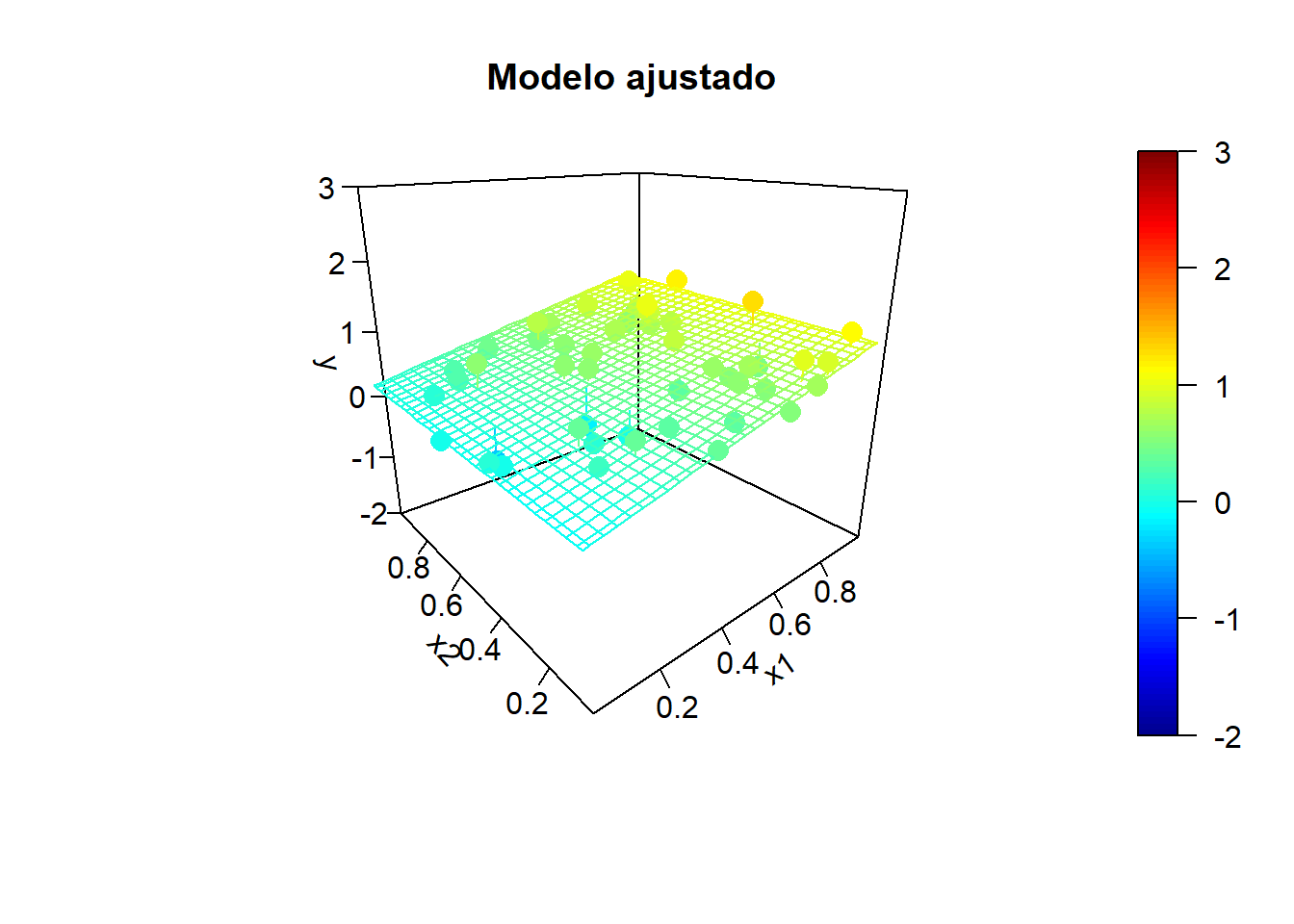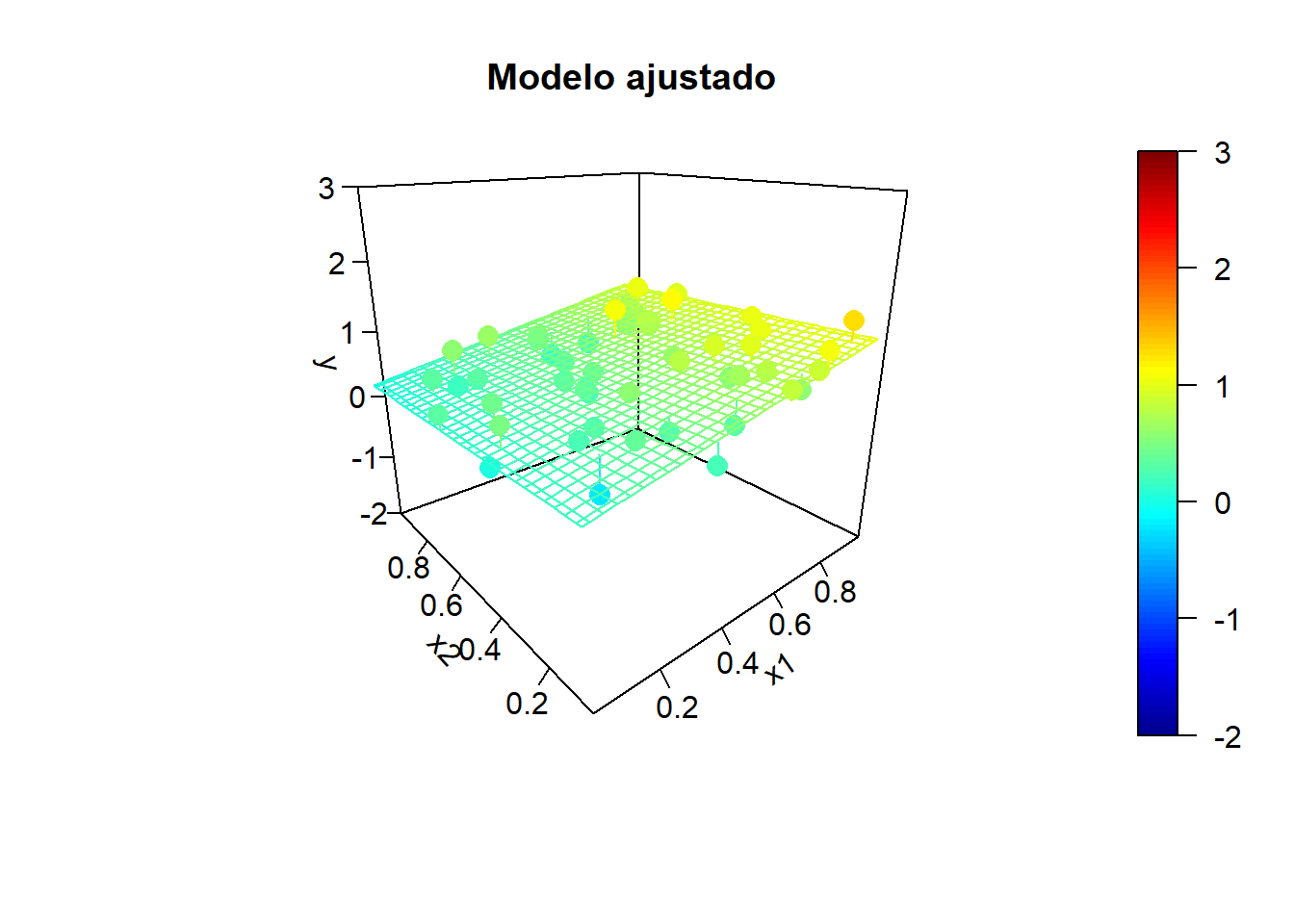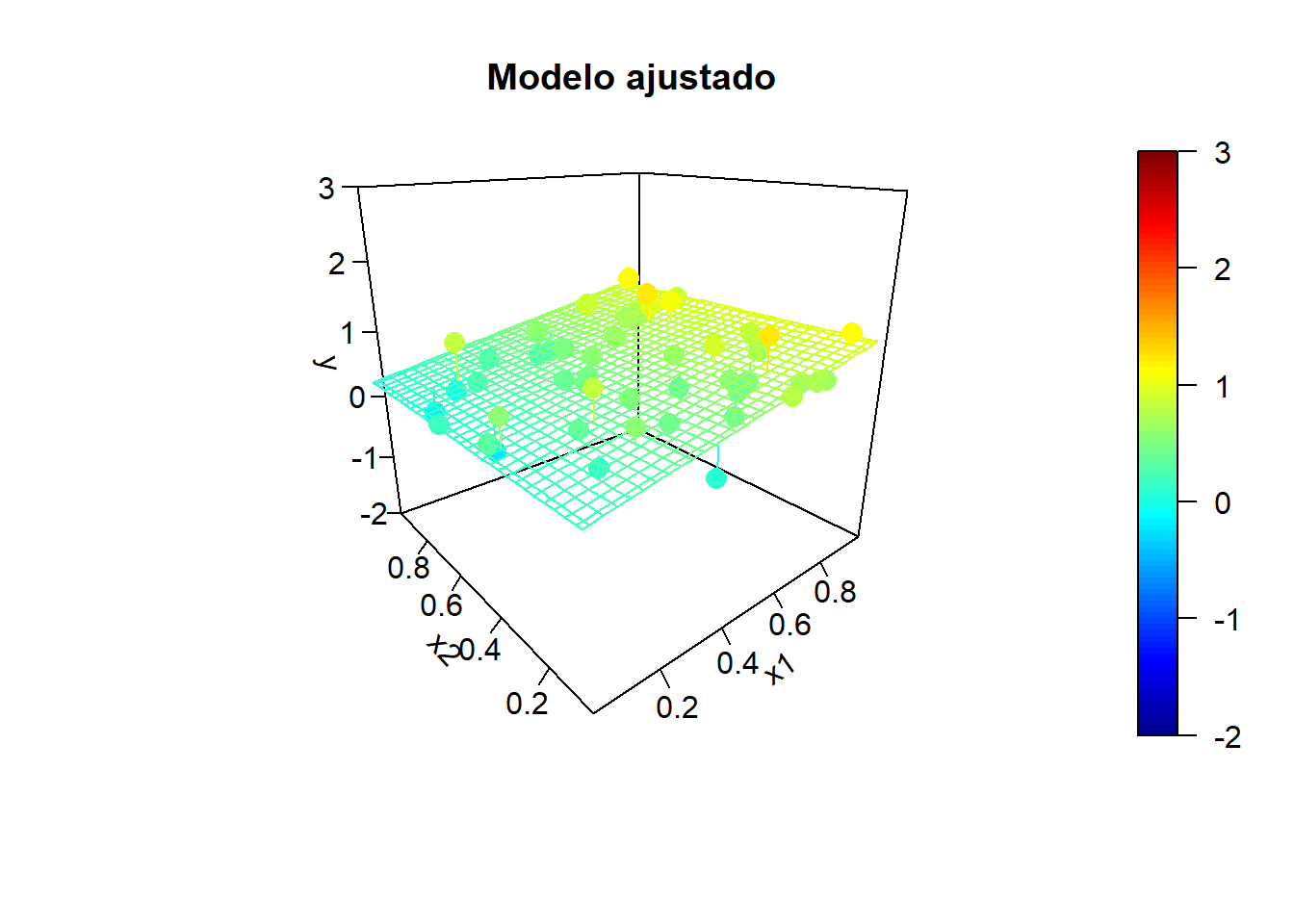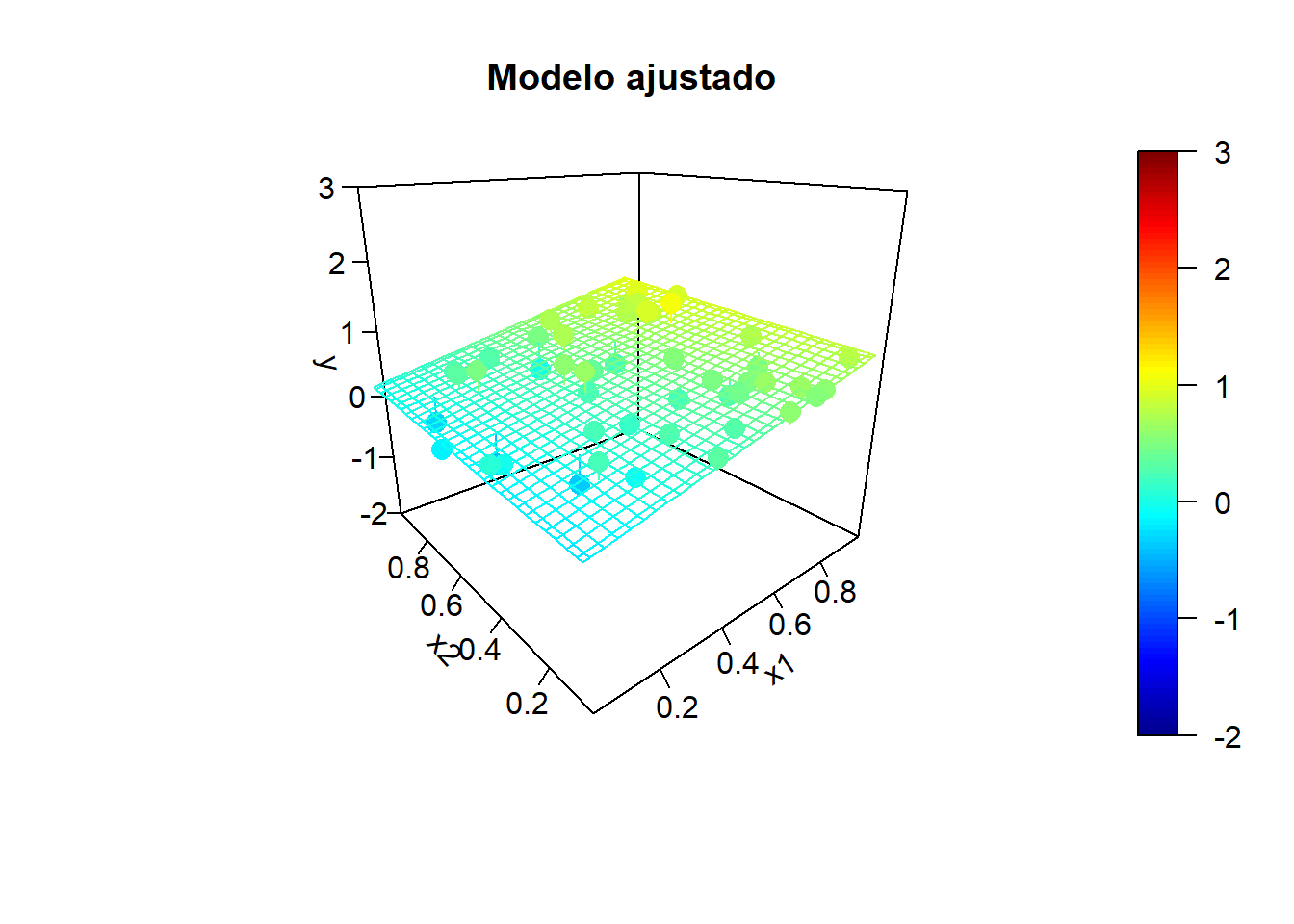
fit <- lm(y ~ x1 + x2)
y.pred <- matrix(predict(fit, newdata = xy), nrow = length(x1.grid))
# Representar
fitpoints <- predict(fit)
scatter3D(z = y, x = x1, y = x2, pch = 16, cex = 1.5, clim = ylim, zlim = ylim,
theta = -40, phi = 20, ticktype = "detailed",
main = "Modelo ajustado", xlab = "x1", ylab = "x2", zlab = "y",
surf = list(x = x1.grid, y = x2.grid, z = y.pred,
facets = NA, fit = fitpoints))
}
Incluso puede ocurrir que el contraste de regresión sea significativo (alto coeficiente de determinación), pero los contrastes individuales sean no significativos.
Por ejemplo, en el último ajuste obtendríamos:
summary(fit)##
## Call:
## lm(formula = y ~ x1 + x2)
##
## Residuals:
## Min 1Q Median 3Q Max
## -0.45461 -0.13147 0.01428 0.16316 0.36616
##
## Coefficients:
## Estimate Std. Error t value Pr(>|t|)
## (Intercept) -0.11373 0.08944 -1.272 0.210
## x1 0.87084 1.19929 0.726 0.471
## x2 0.16752 1.19337 0.140 0.889
##
## Residual standard error: 0.2209 on 47 degrees of freedom
## Multiple R-squared: 0.6308, Adjusted R-squared: 0.6151
## F-statistic: 40.15 on 2 and 47 DF, p-value: 6.776e-11Si las variables explicativas no estuviesen correlacionadas:
x2 <- rand.gen(n)
y.mean <- mapply(model.teor, x1, x2)
for (isim in 1:nsim) {
# Simular respuesta
set.seed(isim)
y <- y.mean + rnorm(n, 0, sd.err)
# Ajuste lineal y superficie de predicción
fit2 <- lm(y ~ x1 + x2)
y.pred <- matrix(predict(fit2, newdata = xy), nrow = length(x1.grid))
# Representar
fitpoints <- predict(fit2)
scatter3D(z = y, x = x1, y = x2, pch = 16, cex = 1.5, clim = ylim, zlim = ylim,
theta = -40, phi = 20, ticktype = "detailed",
main = "Modelo ajustado", xlab = "x1", ylab = "x2", zlab = "y",
surf = list(x = x1.grid, y = x2.grid, z = y.pred,
facets = NA, fit = fitpoints))
}
Por ejemplo, en el último ajuste obtendríamos:
summary(fit2)##
## Call:
## lm(formula = y ~ x1 + x2)
##
## Residuals:
## Min 1Q Median 3Q Max
## -0.45800 -0.08645 0.00452 0.15402 0.33662
##
## Coefficients:
## Estimate Std. Error t value Pr(>|t|)
## (Intercept) -0.22365 0.08515 -2.627 0.0116 *
## x1 1.04125 0.11044 9.428 2.07e-12 ***
## x2 0.22334 0.10212 2.187 0.0337 *
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
##
## Residual standard error: 0.2105 on 47 degrees of freedom
## Multiple R-squared: 0.6648, Adjusted R-squared: 0.6505
## F-statistic: 46.6 on 2 and 47 DF, p-value: 7.016e-12En la práctica, para la detección de colinealidad, se puede emplear la función
vif() del paquete car para calcular los factores de inflación de varianza para las variables del modelo.
Por ejemplo, en los últimos ajustes obtendríamos:
library(car)## Loading required package: carDatavif(fit)## x1 x2
## 107.0814 107.0814vif(fit2) ## x1 x2
## 1.000139 1.000139La idea de este estadístico es que la varianza de la estimación del efecto en regresión simple (efecto global) es menor que en regresión múltiple (efecto parcial). El factor de inflación de la varianza mide el incremento debido a la colinealidad. Valores grandes, por ejemplo > 10, indican la posible presencia de colinealidad.
Las tolerancias, proporciones de variabilidad no explicada por las demás covariables, se pueden calcular con 1/vif(modelo).
Por ejemplo, los coeficientes de tolerancia de los últimos ajustes serían:
1/vif(fit)## x1 x2
## 0.009338689 0.0093386891/vif(fit2) ## x1 x2
## 0.9998606 0.9998606El problema de la colinealidad se agrava al aumentar el número de dimensiones (la maldición de la dimensionalidad). Hay que tener en cuenta también que, además de la dificultad para interpretar el efecto de los predictores, va a resultar más difícil determinar que variables son de interés para predecir la respuesta (i.e. no son ruido). Debido a la aleatoriedad, predictores que realmente no están relacionados con la respuesta pueden ser tenidos en cuenta por el modelo con mayor facilidad, especialmente si se recurre a los contrastes tradicionales para determinar si tienen un efecto significativo.
Por ejemplo en el último ajuste, bajo las hipótesis del modelo de regresión lineal múltiple, se aceptaría un efecto lineal significativo de x2 aunque realmente no influye en la respuesta (esto puede ocurrir el 5% de las veces, considerando una única variable).