Introducción
Repaso: La covarianza y el coeficiente de correlación
Para medir la relación (lineal) entre dos variables se emplea la covarianza: \[Cov(X,Y) = E{\big[(X - E(X))(Y - E(Y))\big]}\] Su estimador es la covarianza muestral: \[s_{XY}=\frac{1}{n}\sum_{i=1}^{n}(x_{i}-\overline{x})(y_{i} - \overline{y})\]
Si dos variables son idependientes su covarianza es nula. El reciproco no es cierto en general, si dos variables tienen covarianza nula se dice que son incorreladas (no hay relación lineal, aunque puede haber una relación no lineal).
Si la covarianza es positiva indica que a valores grandes de X le corresponden valores grandes de Y (i.e. al incrementar X se incrementa Y) y se dice que hay una relación lineal positiva.
Si la covarianza es negativa indica que a valores grandes de X le corresponden valores pequeños de Y (i.e. al incrementar X, Y disminuye) y se dice que hay una relación lineal negativa.
Cuanto mayor es el valor (absoluto) de la covarianza, mayor es el grado de relación lineal entre las variables. Sin embargo, su valor depende de las escala de las variables por lo que es difícil determinar cuando es grande o pequeña. Para medir el grado de relación lineal puede ser preferible reescalarla, i.e. emplear el coeficiente de correlación: \[\rho \left( X, Y \right) =\frac{ Cov \left( X, Y \right) } { \sigma \left( X \right) \sigma \left( Y \right) }\]
Su estimador es el coeficiente de correlación muestral: \[r_{XY}=\frac{\sum_{i=1}^{n}(x_i-\overline{x})(y_i-\overline{y})} {\sqrt{ \sum_{i=1}^{n}(x_i-\overline{x})^{2}} \sqrt{\sum_{i=1}^{n}(y_i-\overline{y})^{2}}}\]
Matriz de covarianzas
Cuando consideramos \(d\) variables aleatorias,
\[\mathbf{Y} = \left( Y_{1},Y_{2},\ldots ,Y_{d}\right),\]
se trabaja con la matriz de covarianzas:
\[\Sigma = E{\big[(\mathbf{Y} - E(\mathbf{Y}))(\mathbf{Y} - E(\mathbf{Y}))^t \big]}\]
donde \(\sigma_{ij} = Cov(Y_i,Y_j)\)
(también se denomina matriz de varianzas-covarianzas)
Ejemplo: datos simulados
Consideramos un proceso temporal estacionario con dependencia exponencial:
curve(exp(-x), 0, 2, xlab = 'distancia', ylab = 'covarianza', ylim = c(0,1))
abline(h = 0, v = 1, lty = 3)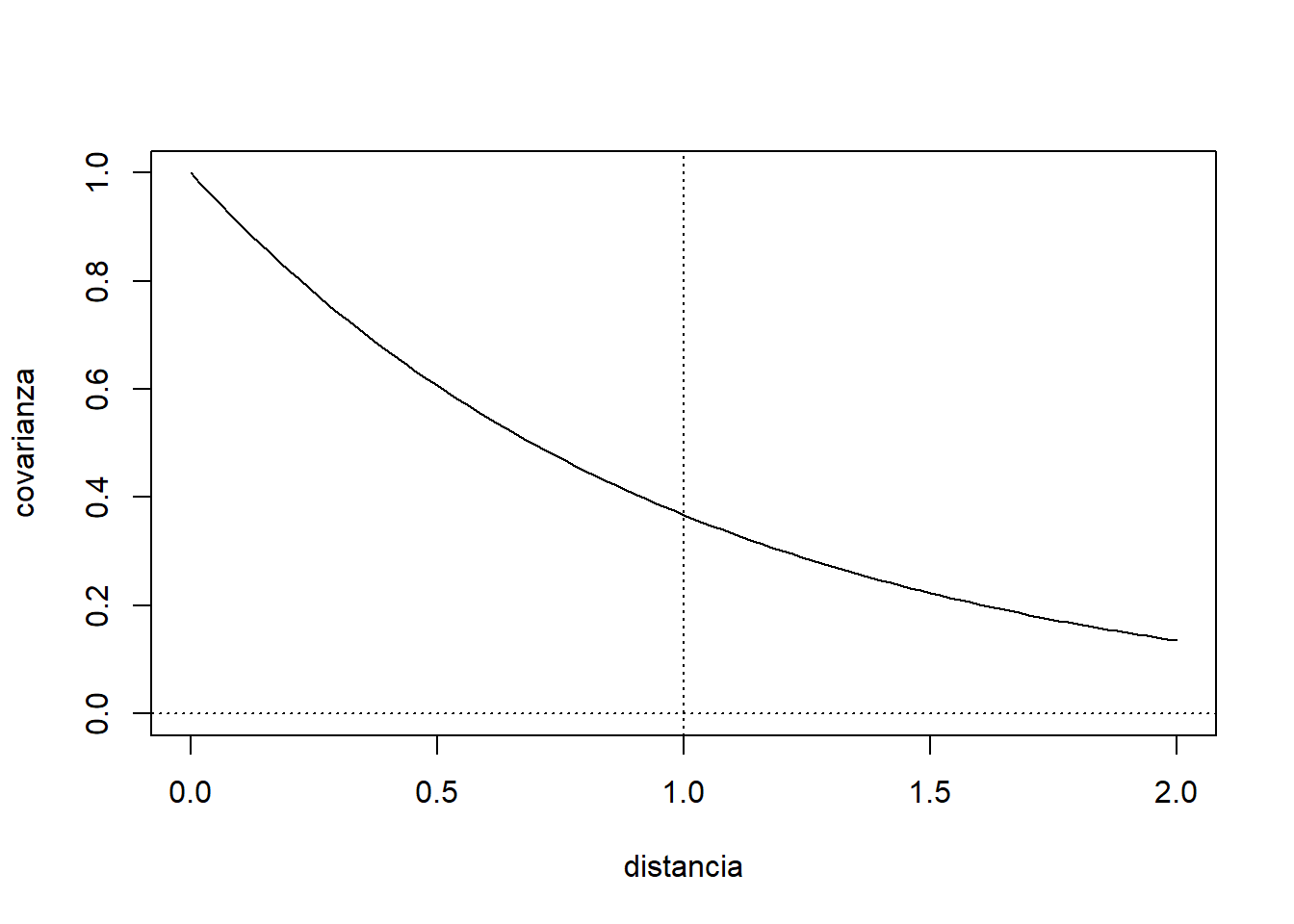
(la dependencia entre las observaciones depende del “salto” entre ellas).
n <- 100 # Nº de observaciones
t <- seq(0, 1, length = n)
mu <- rep(0, n) # Media
# mu <- 0.25 + 0.5*t
# mu <- sin(2*pi*t)
# Matriz de covarianzas
t.dist <- as.matrix(dist(t))
t.cov <- exp(-t.dist)
# str(t.cov)
# num [1:100, 1:100] 1 0.99 0.98 0.97 0.96 ...
t.cov[1:5, 1:5] # Covarianzas de las 5 primeras observaciones # 1 2 3 4 5
# 1 1.0000000 0.9899498 0.9800007 0.9701515 0.9604013
# 2 0.9899498 1.0000000 0.9899498 0.9800007 0.9701515
# 3 0.9800007 0.9899498 1.0000000 0.9899498 0.9800007
# 4 0.9701515 0.9800007 0.9899498 1.0000000 0.9899498
# 5 0.9604013 0.9701515 0.9800007 0.9899498 1.0000000# Simulación de las observaciones
set.seed(1)
library(MASS)
z <- rnorm(n)
y1 <- mu + z # Datos independientes
y2 <- mvrnorm(1, mu, t.cov) # Datos dependientes
plot(t, mu, type="l", lwd = 2, ylim = c(-3,3), ylab = 'y')
lines(t, y1, col = 'blue')
lines(t, y2, col = 'red')
legend("bottomright", legend = c("Datos independientes", "Datos dependientes"), col = c('blue', 'red'), lty = 1)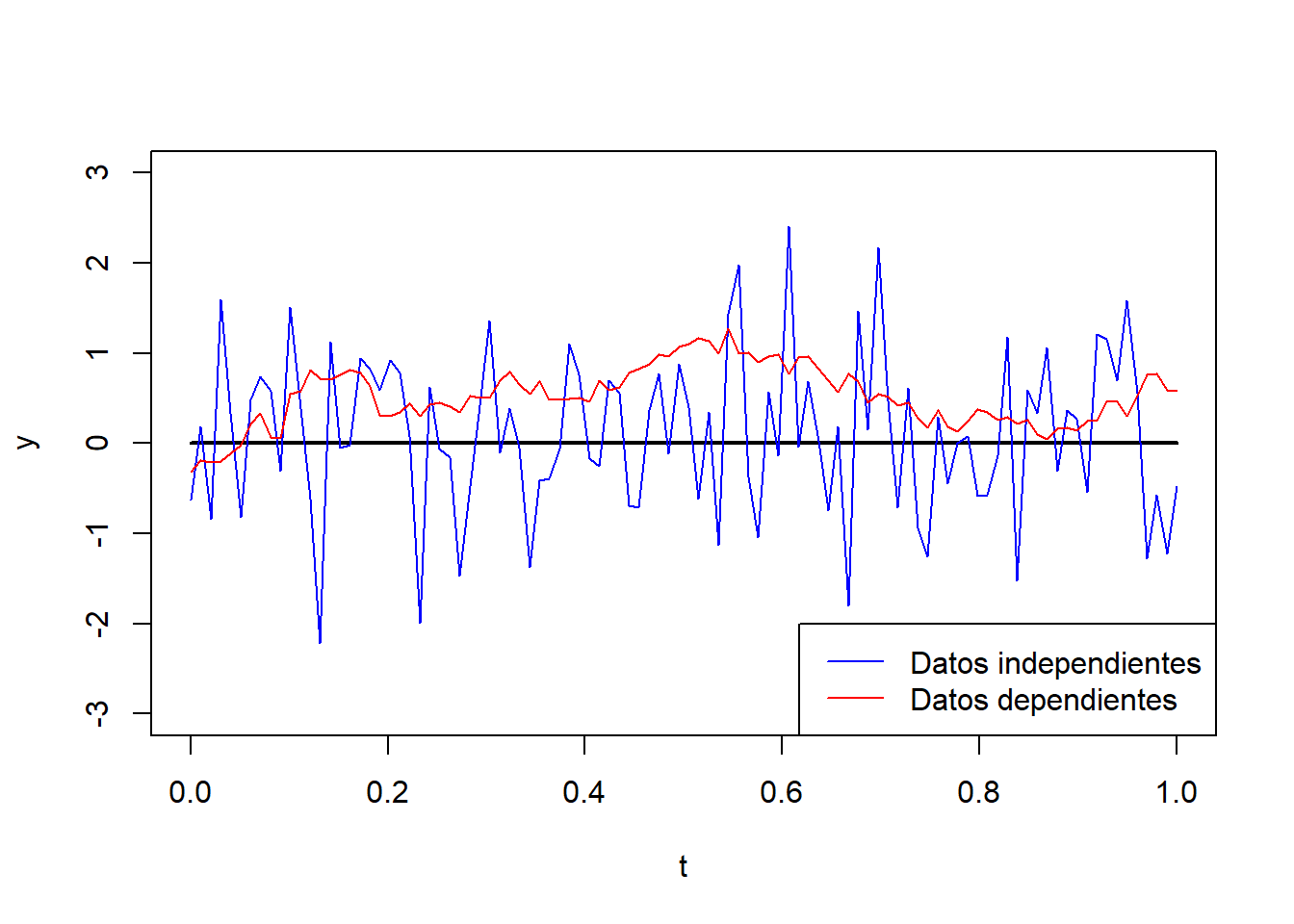
El problema de la dependencia
Los métodos “clásicos” de inferencia estadística se basan en suponer que las observaciones \(Y_{1},\ldots,Y_{n}\) son una muestra aleatoria simple (m.a.s.) de \(Y\). Por tanto suponen que las observaciones son independientes (o los errores, en el caso de un modelo de regresión).
La ausencia de aleatoriedad es difícil de corregir y puede influir notablemente en el análisis estadístico.
Si existe dependencia entre las observaciones muestrales (i.e. el conocimiento de \(Y_{i}\) proporciona información sobre los valores de \(Y_{i+1}\), \(Y_{i+2}\), \(\ldots\)), los métodos “clásicos” no serán en principio adecuados (pueden conducir a conclusiones erróneas).
Esto es debido principalmente a que introduce un sesgo en los estimadores de las varianzas (obtenidos asumiendo independencia).
Los correspondientes intervalos de confianza y contrastes de hipótesis tendrán una confianza o una potencia distinta de la que deberían (aunque las estimaciones de los parámetros pueden no verse muy afectadas).
Si \(Y_{1}\) e \(Y_{2}\) son independientes (\(Cov(Y_{1},Y_{2})=0\)): \[Var(Y_{1}+Y_{2})=Var(Y_{1})+Var(Y_{2})\]
En el caso general (dependencia): \[Var(Y_{1}+Y_{2})=Var(Y_{1})+Var(Y_{2})+2Cov(Y_{1},Y_{2})\]
Típicamente \(Cov(Y_{1},Y_{2})>0\) por lo que con los métodos “clásicos” (basados en independencia) se suelen producir subestimaciones de las varianzas (IC más estrechos y tendencia a rechazar \(H_{0}\) en contrastes).
Ejemplo: datos simulados
En el caso anterior la varianza es uno con ambos procesos. Las estimaciones suponiendo independencia serían:
var(y1) # [1] 0.8067621var(y2) # [1] 0.1108155En el caso de datos dependientes se produce una clara subestimación de la varianza
Métodos para detectar dependencia
Es de esperar que datos cercanos en el tiempo (o en el espacio) sean más parecidos (dependientes) que datos más alejados \(\Rightarrow\) dependencia temporal (espacial, espacio-temporal).
En este tema nos centraremos en el caso de dependencia temporal (unidimensional), más adelante trataremos el caso de dependecia espacial (bidimensional).
Métodos para detectar dependencia temporal
Gráficos:
Secuencial / Dispersión frente al tiempo
Dispersión retardado
Correlograma
Contrastes:
Tests basados en rachas
Test de Ljung-Box
Métodos gráficos
Gráfico secuencial
El gráfico de dispersión \(\{(i,Y_{i}) : i = 1, \ldots, n \}\) permite detectar la presencia de un efecto temporal.
Es importante mantener/guardar el orden de recogida de los datos.
Si existe una tendencia los datos no son homogéneos (debería tenerse en cuenta la variable índice, o tiempo, como variable explicativa). Podría indicar la presencia de un “efecto aprendizaje”.
Comandos R:
plot(as.ts(y))
Ejemplo
Datos mensuales de CO2 (ppm) en el ambiente entre enero de 1990 y diciembre de 1997
load("data/co2.RData")
t <- seq(1990, by=1/12, length=length(co2$y))
y <- co2$y
plot(as.ts(y))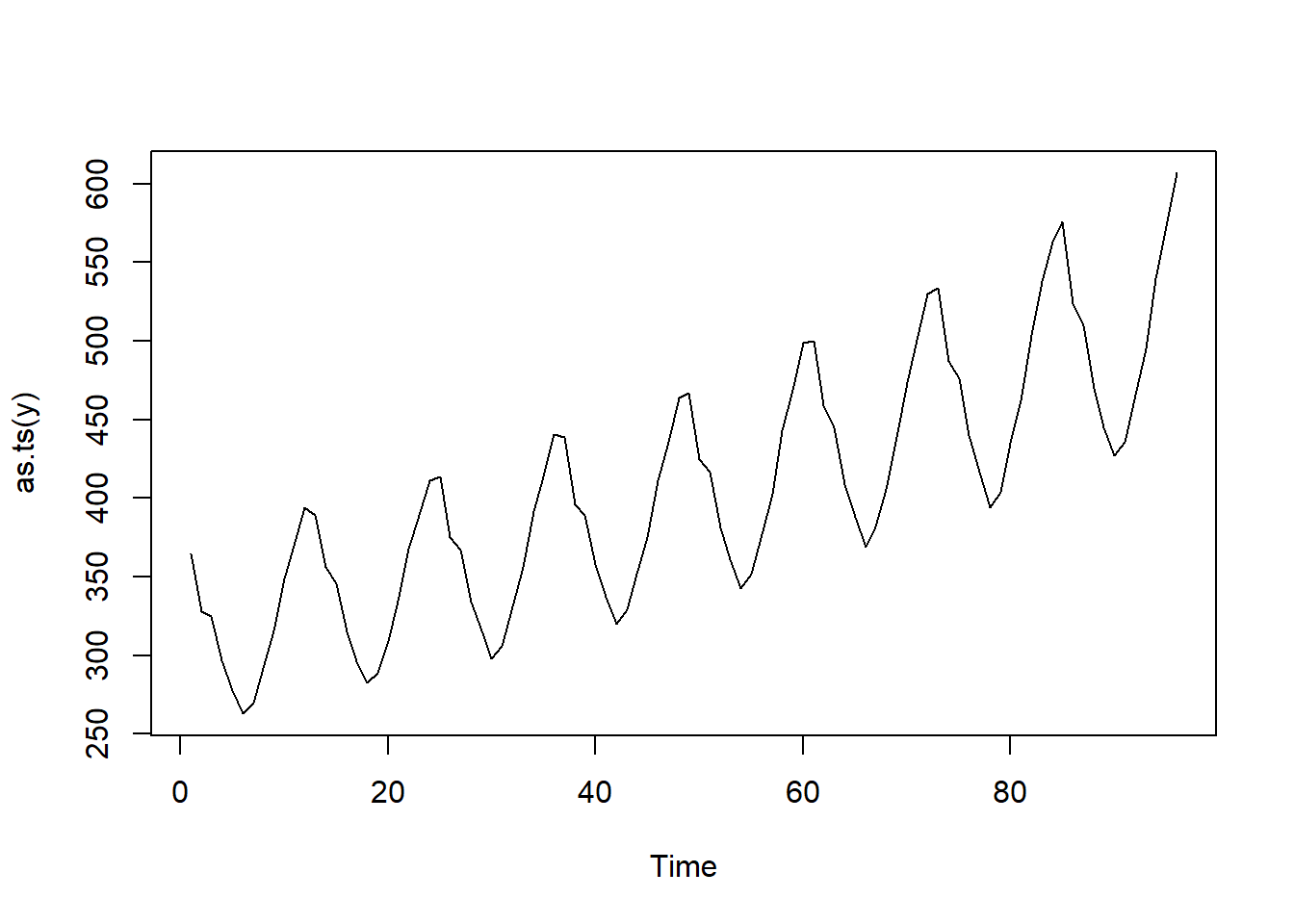
Es habitual que este tipo de análisis se realice sobre los residuos
de un modelo de regresión (e.g. datos <- residuals(modelo))
modelo <- lm(y ~ t, data = co2)
plot(y ~ t, data = co2)
abline(modelo)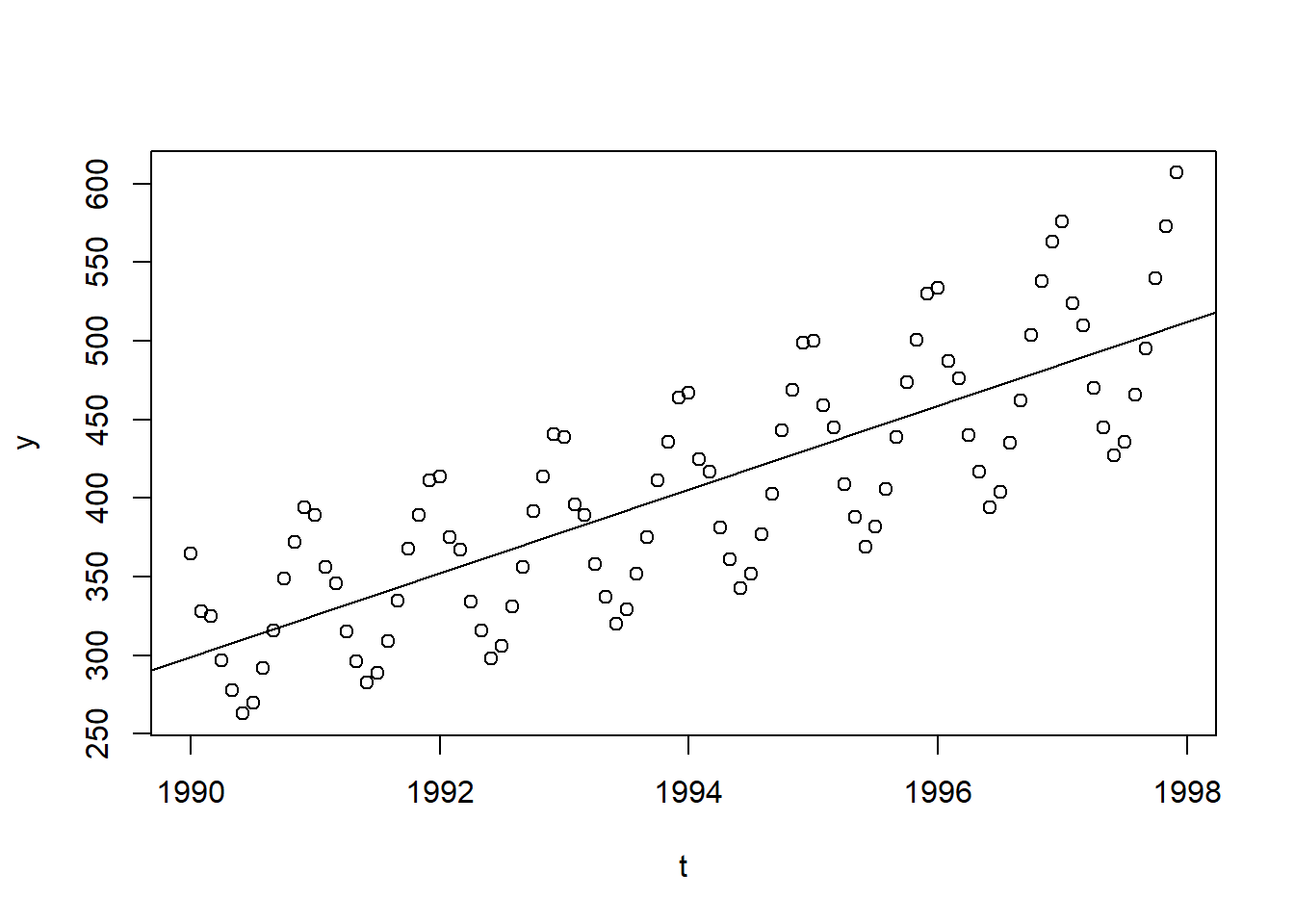
r <- residuals(modelo)
plot(as.ts(r))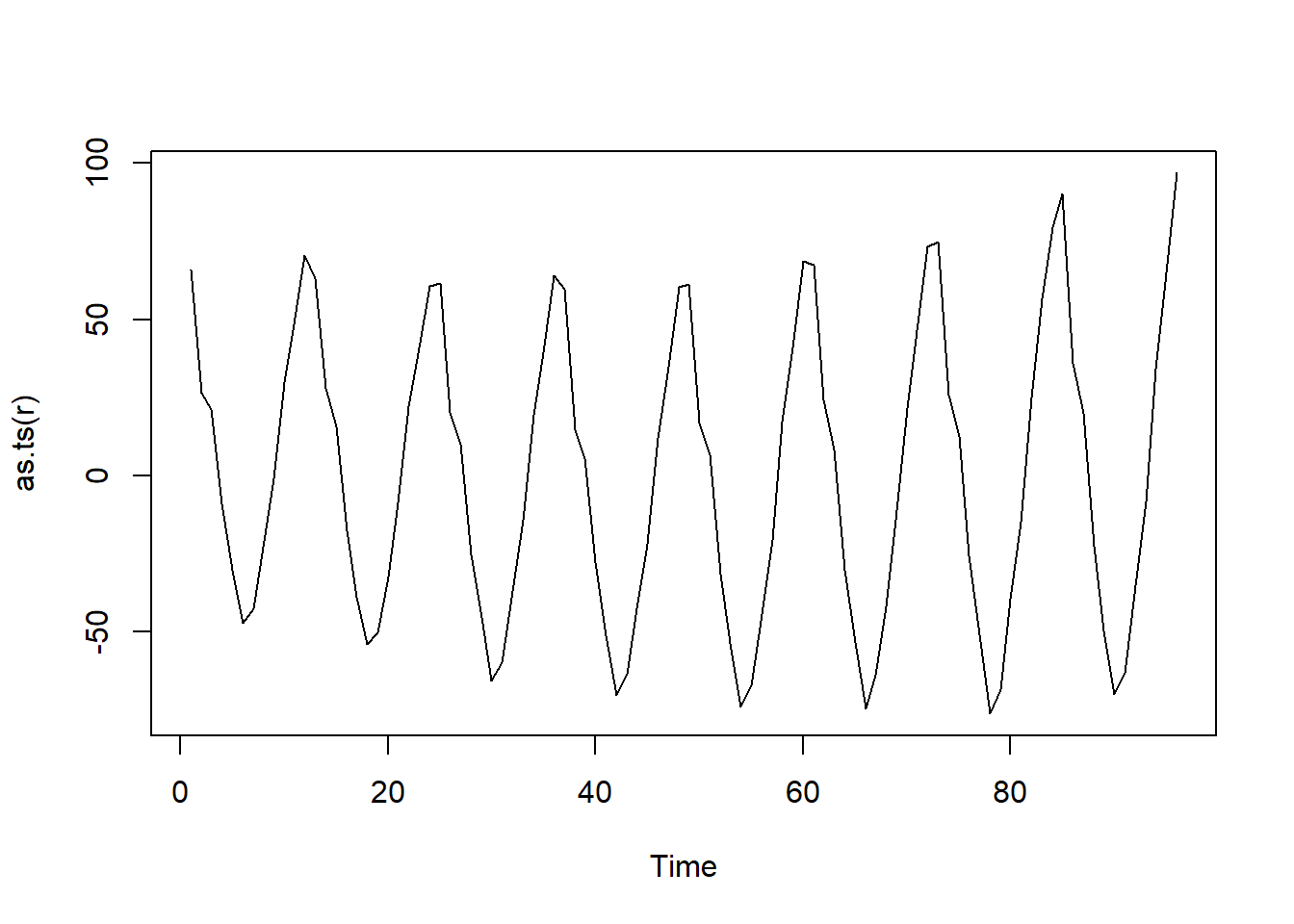
En este caso se observa una componente estacional (anual), habría que incluirla en la tendencia. Por ejemplo:
modelo <- lm(y ~ t + as.factor(month), data = co2)
plot(y ~ t, data = co2)
lines(t, predict(modelo))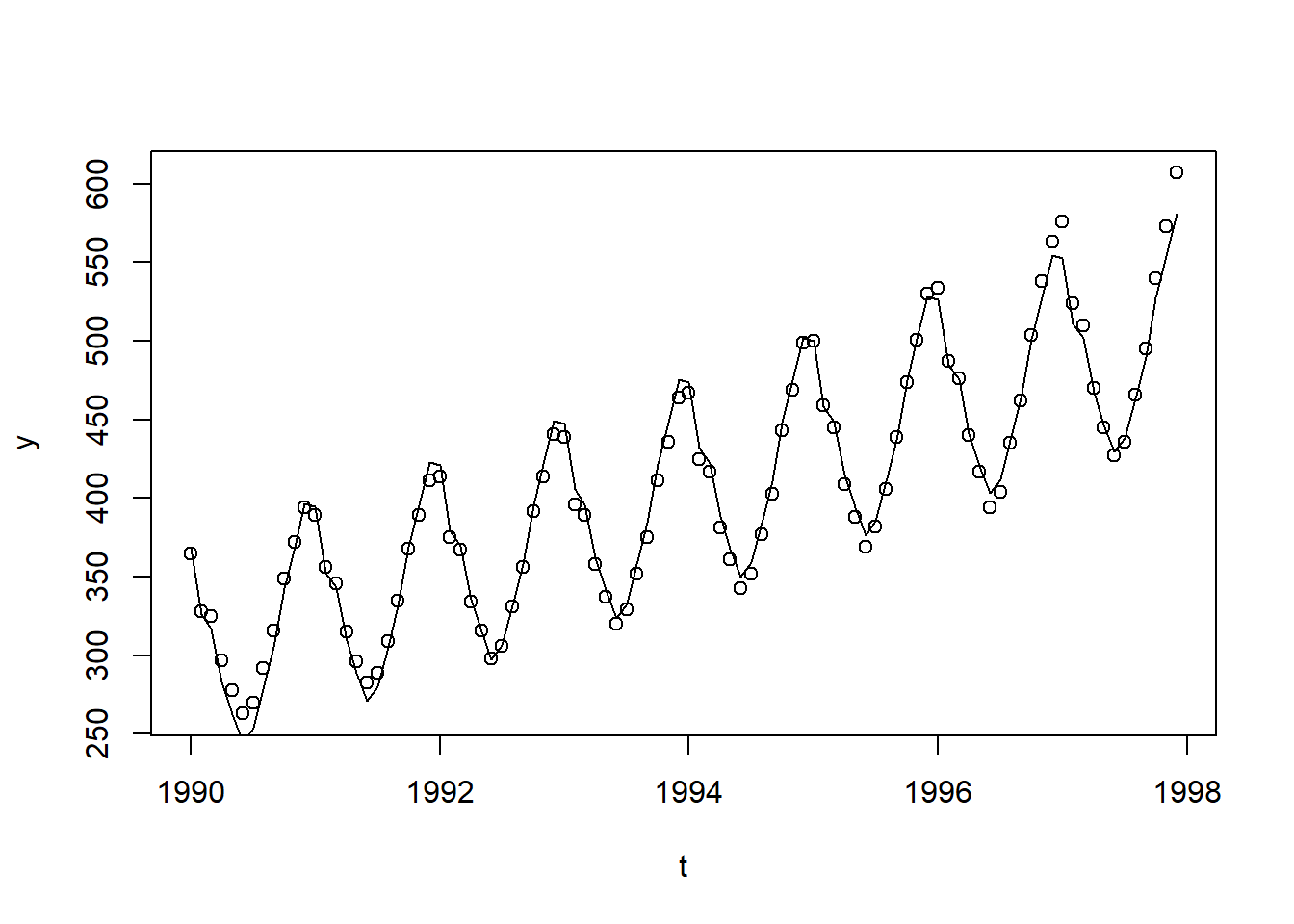
r <- residuals(modelo)
plot(as.ts(r))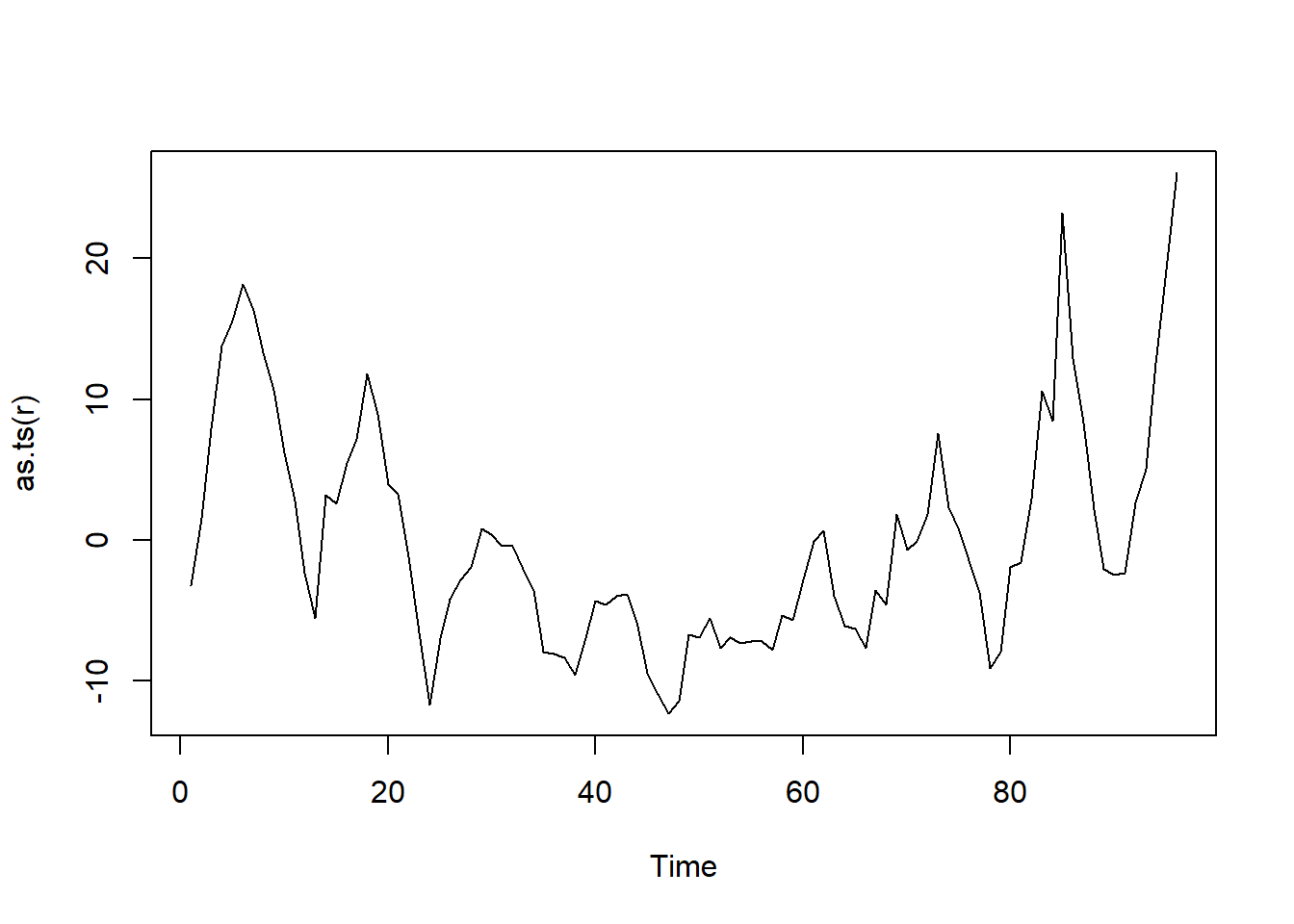
Dependencia
Este gráfico también podría servir para detectar dependencia temporal:
Valores próximos muy parecidos (valores grandes seguidos de grandes y viceversa) indicarían una posible dependencia positiva.
Valores próximos dispares (valores grandes seguidos de pequeños y viceversa) indicarían una posible dependencia negativa.
old.par <- par(mfrow = c(1, 3))
plot(y2, type = 'l', ylab = '', main = 'Dependencia positiva')
plot(y1, type = 'l', ylab = '', main = 'Independencia')
y3 <- y2 * c(1, -1)
plot(y3, type = 'l', ylab = '', main = 'Dependencia negativa')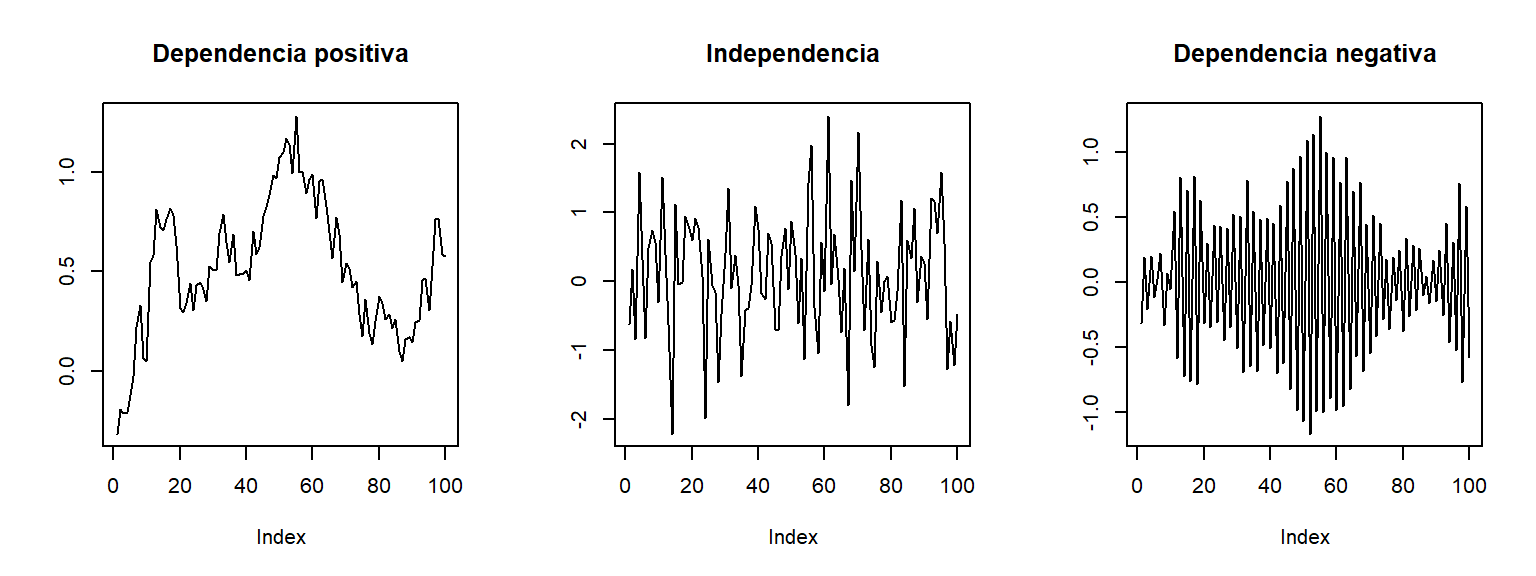
par(old.par)pero suele ser preferible emplear un gráfico de dispersión retardado.
Gráfico de dispersion retardado
El gráfico de dispersión \(\{(Y_{i},Y_{i+1}) : i = 1, \ldots, n-1 \}\) permite detectar dependencias a un retardo (relaciones entre valores separados por un instante)
- Comando R:
plot(y[-length(y)], y[-1], xlab = "Y_t", ylab = "Y_t+1")
old.par <- par(mfrow = c(1, 3))
plot(y2[-length(y2)], y2[-1], xlab = "Y_t", ylab = "Y_t+1", main = 'Dependencia positiva')
plot(y1[-length(y1)], y1[-1], xlab = "Y_t", ylab = "Y_t+1", main = 'Independencia')
plot(y3[-length(y3)], y3[-1], xlab = "Y_t", ylab = "Y_t+1", main = 'Dependencia negativa')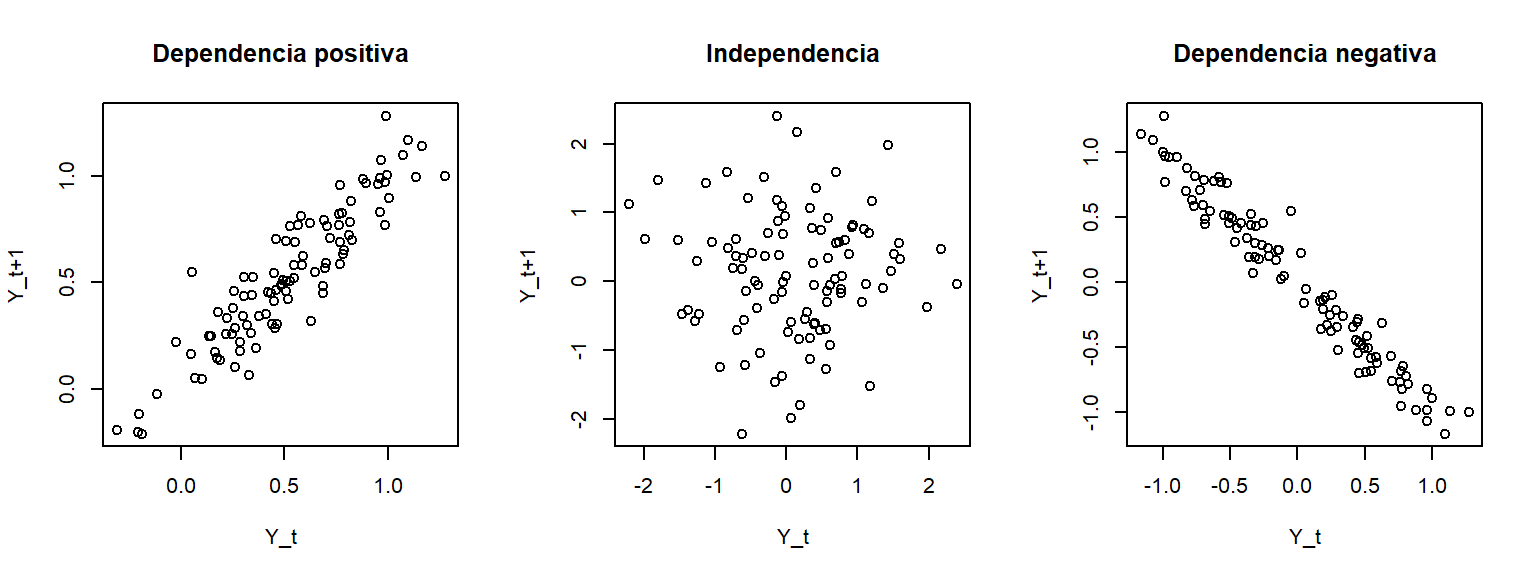
par(old.par)Se puede generalizar al gráfico \(\{(Y_{i},Y_{i+k}) : i = 1, \ldots, n-k \}\) que permite detectar dependencias a \(k\) retardos (separadas \(k\) instantes).
Ejemplo: residuos mediciones co2
# Gráfico de dispersion retardado
plot(r[-length(r)], r[-1], xlab = "Y_t", ylab = "Y_t+1")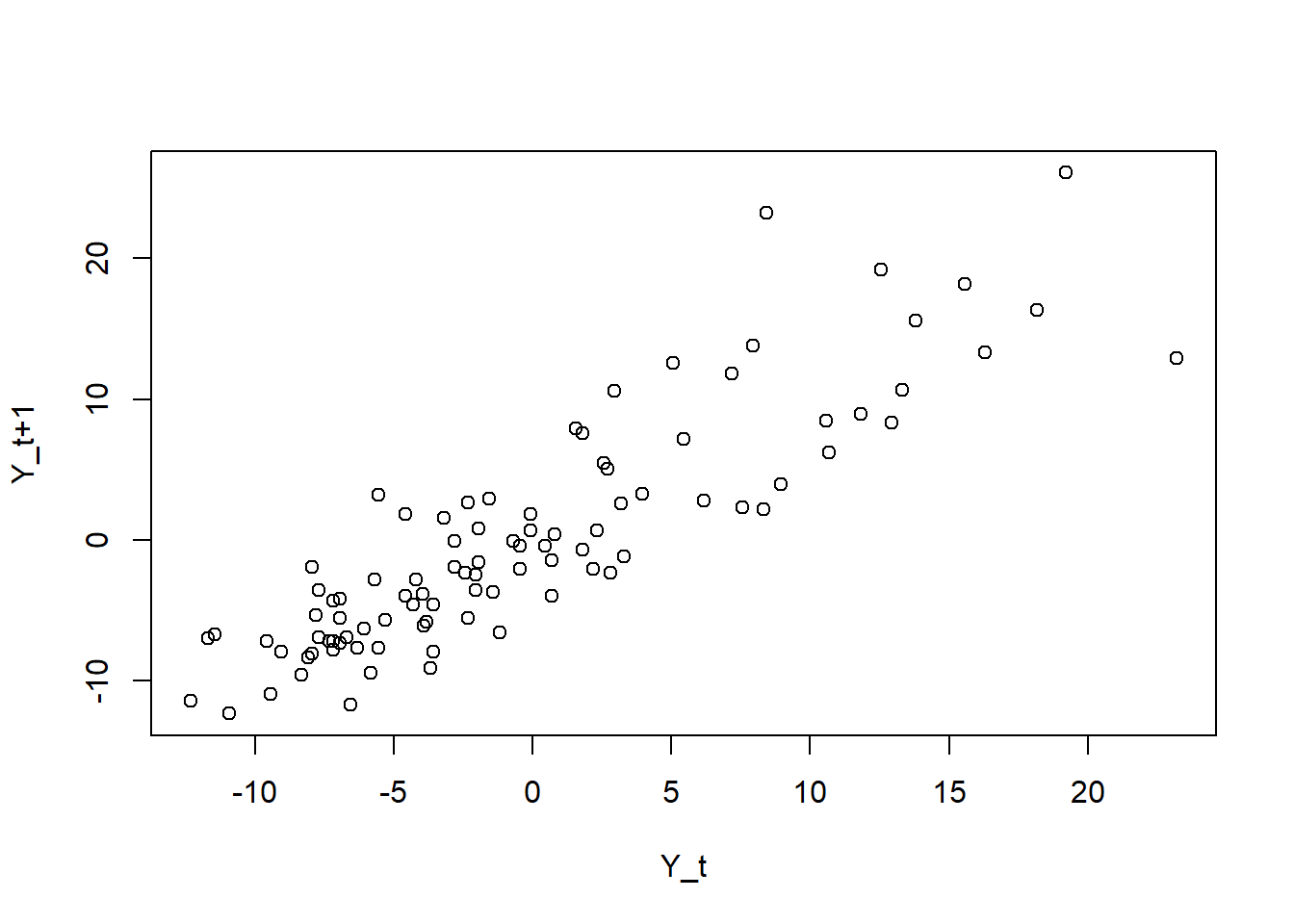
El correspondiente coeficiente de correlación es una medida numérica del grado de relación lineal (denominado autocorrelación de orden 1).
cor(r[-length(r)], r[-1]) # [1] 0.8768295El correlograma
Para estudiar si el grado de relación (lineal) entre \(Y_{i}\) e \(Y_{i+k}\) podemos utilizar el coeficiente de correlación:
\[\rho\left( Y_{i},Y_{i+k}\right) =\frac{Cov\left( Y_{i},Y_{i+k}\right) } {\sigma\left( Y_{i}\right) \sigma\left( Y_{i+k}\right) }\]
En el caso de datos homogéneos (estacionarios): \[\rho\left( Y_{i},Y_{i+k}\right) \equiv\rho\left( k\right)\] denominada función de autocorrelación simple (fas) o correlograma.
Su estimador es el correlograma muestral: \[r(k)=\frac{\sum_{i=1}^{n-k}(Y_{i}-\overline{Y})(Y_{i+k}-\overline{Y})} {\sum_{i=1}^{n}(Y_{i}-\overline{Y})^{2}}\]
Comando R:
acf(y)
En caso de independencia es de esperar que las autocorrelaciones muestrales sean próximas a cero (valores “grandes” indicarían dependencia positiva o negativa según el signo).
old.par <- par(mfrow = c(1, 3))
acf(y1, main = 'Independencia')
acf(y2, main = 'Dependencia positiva')
acf(y3, main = 'Dependencia negativa')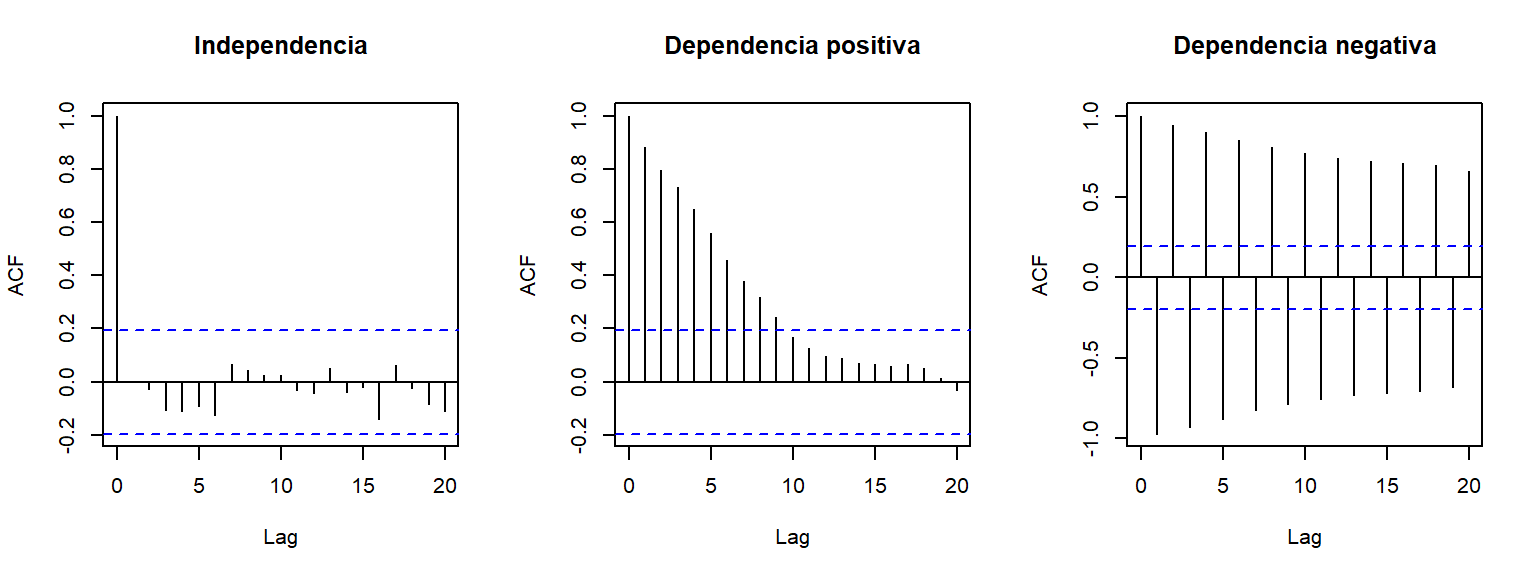
par(old.par)Suponiendo normalidad e independencia, asintóticamente: \[r(k)\underset{aprox.}{\sim}N\left( \rho(k),\frac{1}{n}\right)\]
Si el tamaño muestral es grande, podríamos aceptar \(H_{0}:\) \(\rho\left( k\right) =0\) si:\[|r(k)|<\dfrac{2}{\sqrt{n}}\]
En el gráfico de autocorrelaciones muestrales (también denominado correlograma) se representan las estimaciones \(r(k)\) de las autocorrelaciones correspondientes a los primeros retardos (típicamente \(k<n/4\)) y las correspondientes bandas de confianza (para detectar dependencias significativas).
Ejemplo: residuos mediciones co2
acf(r) # correlaciones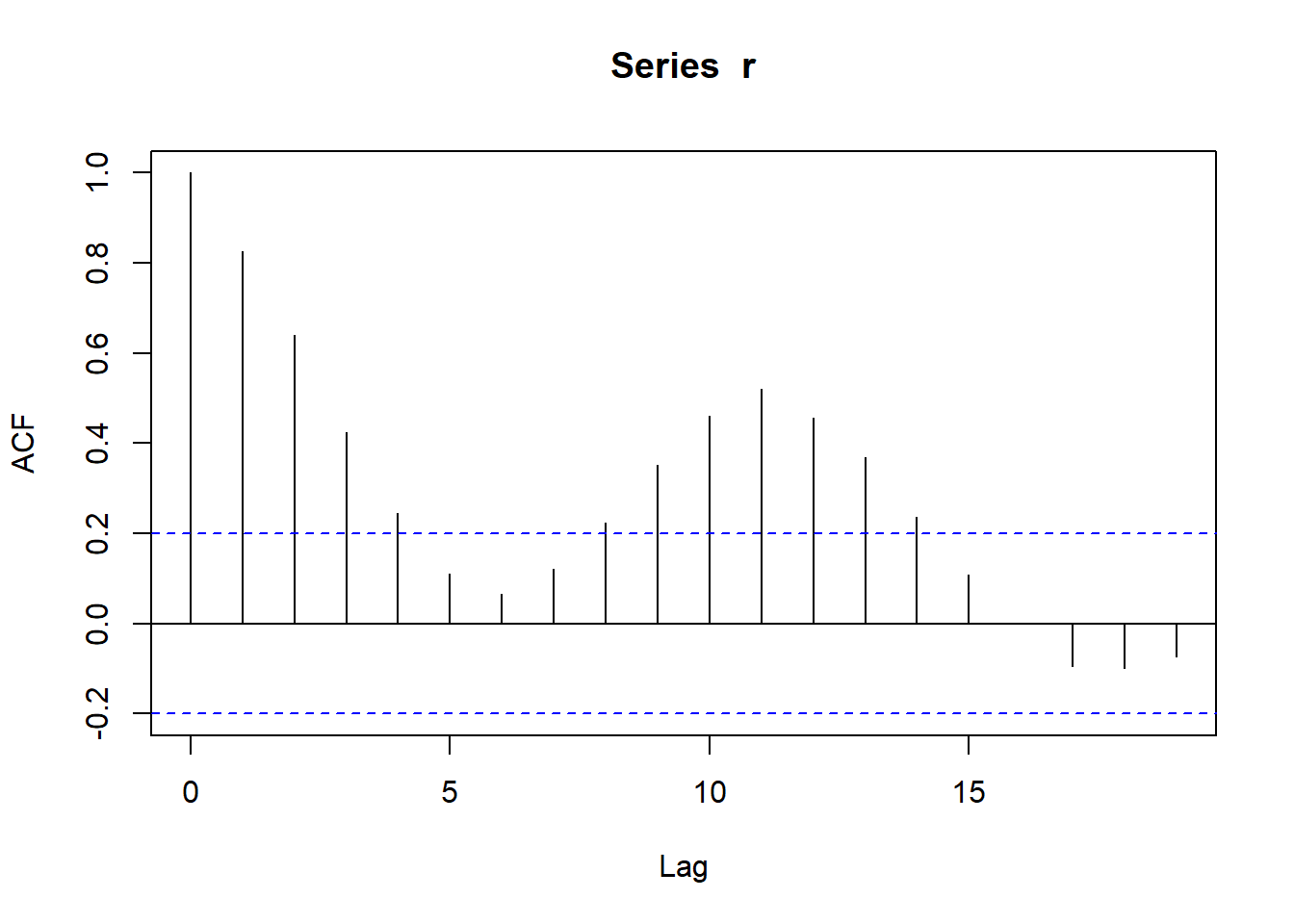
La función acf también permite estimar el covariograma.
covar <- acf(y2, type = "covariance")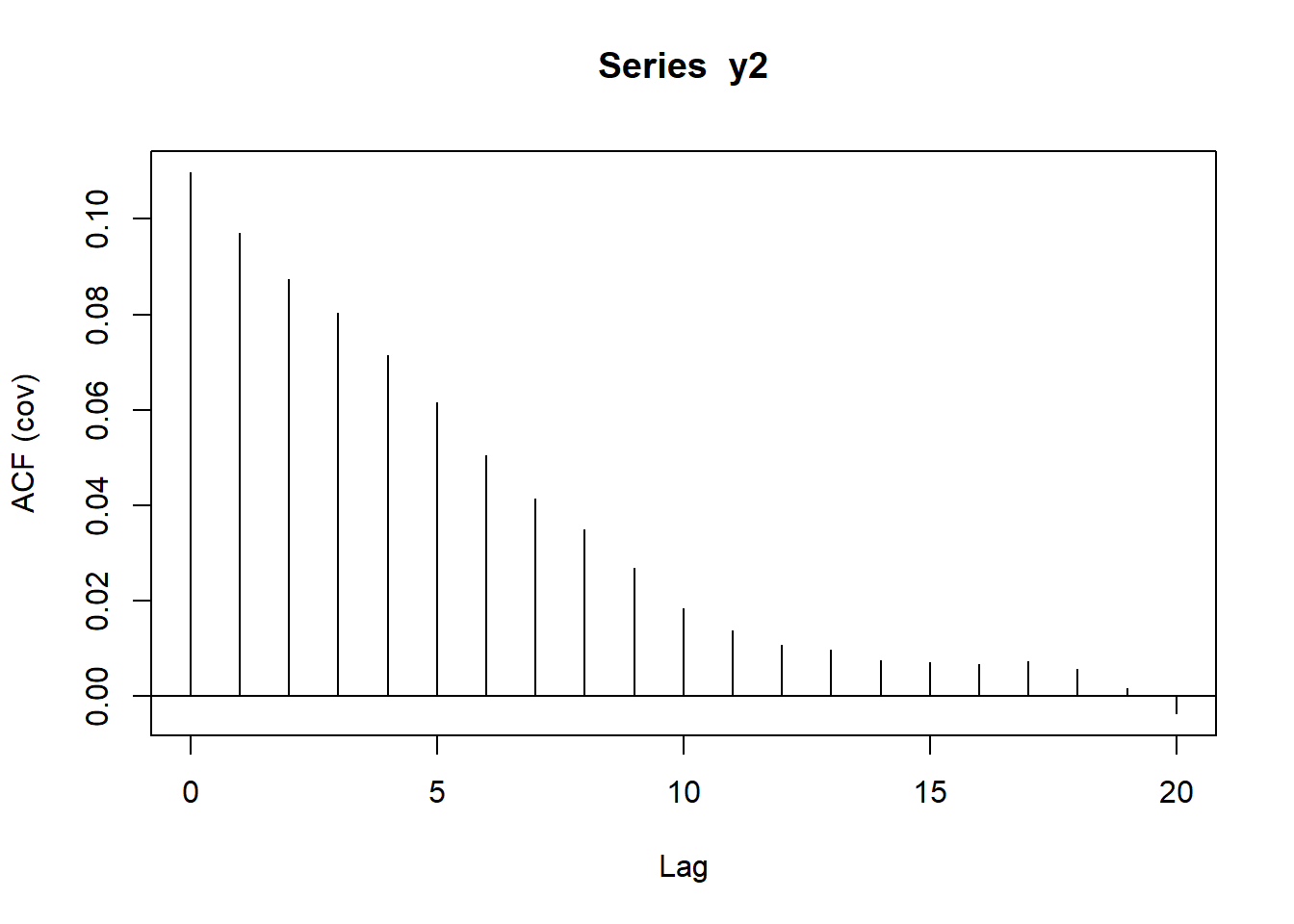
En estadística espacial en lugar del covariograma se suele emplear el semivariograma \(\gamma(k) = C(0) - C(k)\):
plot(covar, type = 'l', ylab = '', main = 'Covariograma y semivariograma (estimados)', col = 'blue')
semivar <- covar$acf[1] - covar$acf
lags <- covar$lag
lines(lags, semivar, col = 'red')
legend("bottomright", legend = c("covariograma", "semivariograma"), col = c('blue', 'red'), lty = 1)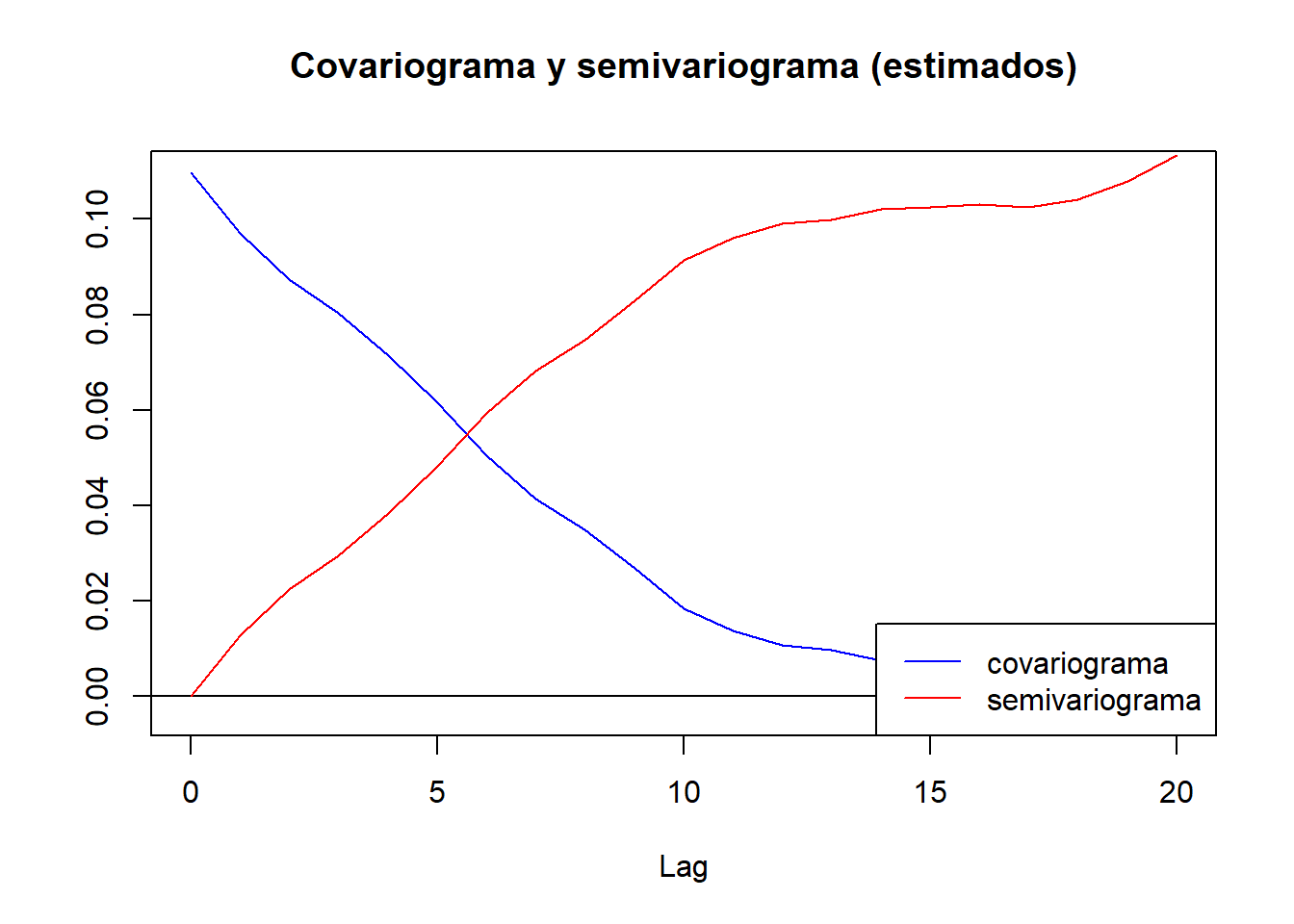
Contrastes de hipótesis
Test de rachas
Permite contrastar si el orden de aparición de dos valores de una variable dicotómica es aleatorio. Supongamos que \(X\) toma los valores \(+\) y \(-\) y que observamos una muestra del tipo: \[++++---+++--++++++----\] y nos interesa contrastar:
\[\left\{ \begin{array}[c]{l} H_{0}:\mathit{La\ muestra\ es\ aleatoria}\\ H_{1}:\mathit{La\ muestra\ no\ es\ aleatoria} \end{array} \right.\]
Una racha es una secuencia de observaciones iguales (o similares): \[\underbrace{++++}_{1}\underbrace{---}_{2}\underbrace{+++}_{3} \underbrace{--}_{4}\underbrace{++++++}_{5}\underbrace{----}_{6}\]
Una muestra con muchas o pocas rachas sugeriría que la muestra no es aleatoria (con dependencia negativa o positiva, respec.).
Estadístico del contraste: \[R=\text{"Nº total de rachas en la muestra"}\]
Bajo la hipótesis nula de aleatoriedad: \[R\underset{aprox.}{\sim}N\left( 1+\frac{2n_{1}n_{2}}{n}, \frac{2n_{1}n_{2}(2n_{1}n_{2}-n)}{n^{2}(n-1)}\right)\] siendo \(n_{1}\) y \(n_{2}\) el nº de signos \(+\) y \(-\) en la muestra, respectivamente (\(n_{1}+n_{2}=n\)). Para tamaños muéstrales pequeños (\(n<40\)), esta aproximación no es buena y conviene utilizar la distribución exacta (o utilizar corrección por continuidad). Los valores críticos de esta distribución están tabulados.
Este contraste se emplea también para variables continuas, se fija un punto de corte para dicotomizarlas. Normalmente se toma como punto de corte la mediana.
En este caso si \(k=n_{1}\) (\(\simeq n_{2}\)): \[R\underset{aprox.}{\sim}N\left( k+1,\frac{k(k-1)}{2k-1}\right)\]
Se rechaza la hipótesis nula de aleatoriedad si el número de rachas es significativamente pequeño o grande.
Si el tamaño muestral es grande, el \(p\)-valor será:\[p\simeq2\cdot P\left( Z\geq\left\vert \frac{R-E(R)}{\sqrt{Var(R)}}\right\vert \right)\]
Comandos R:
library(tseries)
runs.test(as.factor(y > median(y)))Ejemplo: residuos mediciones co2
library(tseries)
runs.test(as.factor(r > median(r))) #
# Runs Test
#
# data: as.factor(r > median(r))
# Standard Normal = -7.1822, p-value = 6.858e-13
# alternative hypothesis: two.sidedEl contraste de Ljung-Box
Es un test muy utilizado (en series de tiempo) para contrastar la hipótesis de independencia.
Se contrasta la hipótesis nula de que las primeras \(m\) autocorrelaciones son cero: \[\left\{\begin{array}[c]{l} H_{0}:\rho_{1}=\rho_{2}=\ldots=\rho_{m}=0\\ H_{1}:\rho_{i}\neq0\text{ para algún } i \end{array} \right.\]
Se elige un \(m\) tal que la estimación \(r(m)\) de \(\rho_{m}=\rho(m)\) sea “fiable” (e.g. \(10\log_{10}n\)).
El estadístico del contraste:\[Q=n(n+2)\sum_{k=1}^{m}\frac{r(k)^{2}}{n-k}\underset{aprox.}{\sim}\chi _{m-1}^{2}\text{, si }H_{0}\text{ es cierta.}\]
Se rechaza \(H_{0}\) si el valor observado es grande (\(Q\geq \chi_{m-1,1-\alpha}^{2}\)):\[p=P\left( {\chi_{m-1}^{2}}\geq Q\right)\]
Comandos R:
Box.test(y, type=Ljung)
Box.test(y, lag, type=Ljung)Ejemplo: residuos mediciones co2
Box.test(r, type="Ljung") # Contrasta si la primera autocorrelación es nula #
# Box-Ljung test
#
# data: r
# X-squared = 67.568, df = 1, p-value = 2.22e-16Box.test(y, lag=10, type="Ljung") # Contrasta si las 10 primeras autocorrelaciones son nulas #
# Box-Ljung test
#
# data: y
# X-squared = 271.5, df = 10, p-value < 2.2e-16NOTA: Para contrastar que la primera autocorrelación es cero, teniendo en cuenta además que los valores son residuos, sería preferible emplear el test de Durbin-Watson visto antes:
library(lmtest)
dwtest(modelo, alternative= "two.sided") #
# Durbin-Watson test
#
# data: modelo
# DW = 0.23671, p-value < 2.2e-16
# alternative hypothesis: true autocorrelation is not 0Ejemplo adicional: Análisis de series temporales
En este caso sería recomendable analizar los datos como una serie temporales. Se trataría de modelar los componentes de la serie de tiempo:
plot(decompose(co2$y.ts))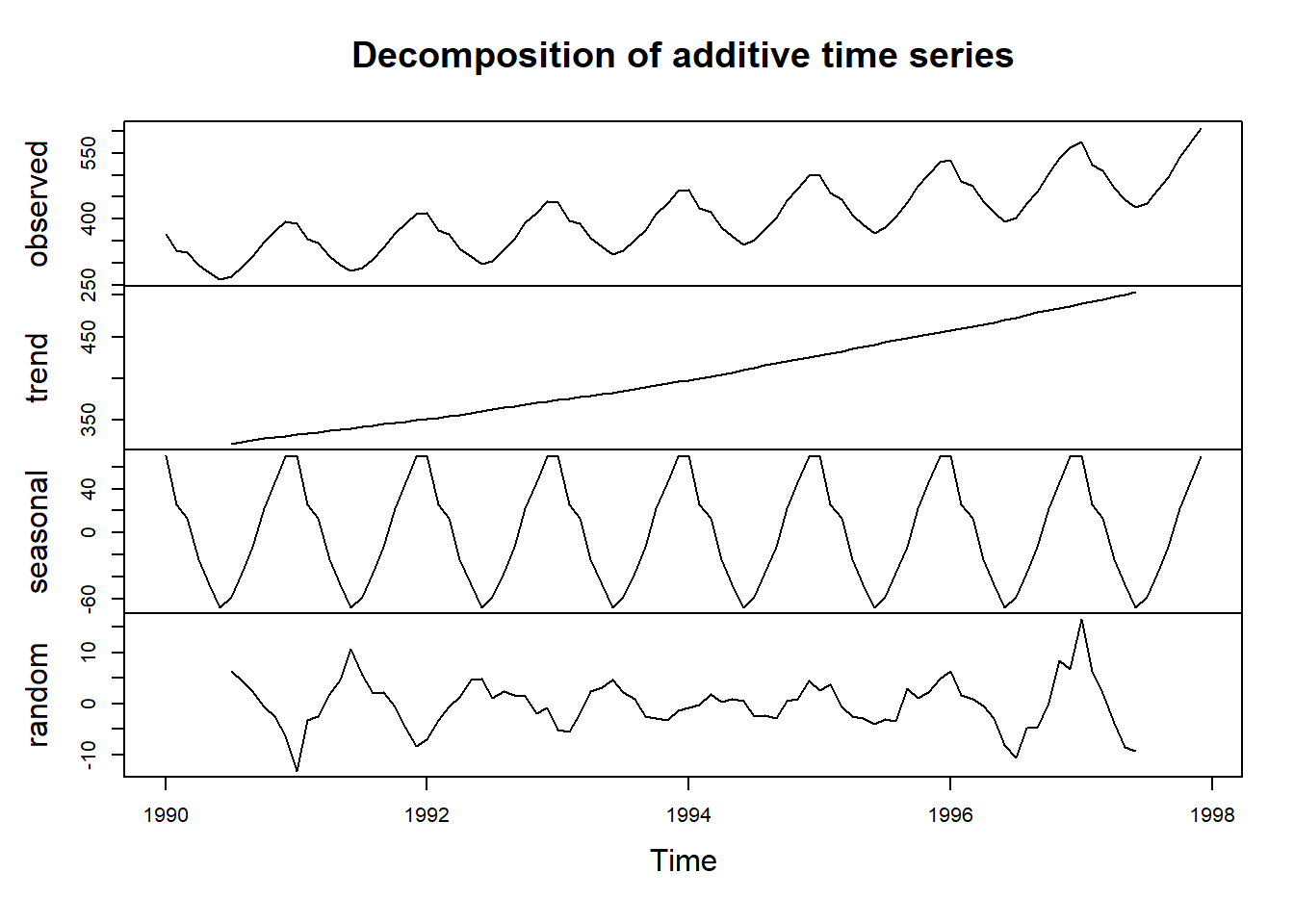
Para ello se suele emplear modelos ARIMA, e.g.:
library(forecast)
fit <- auto.arima(co2$y.ts)
plot(forecast(fit, h = 24, level = 95), shaded = FALSE) 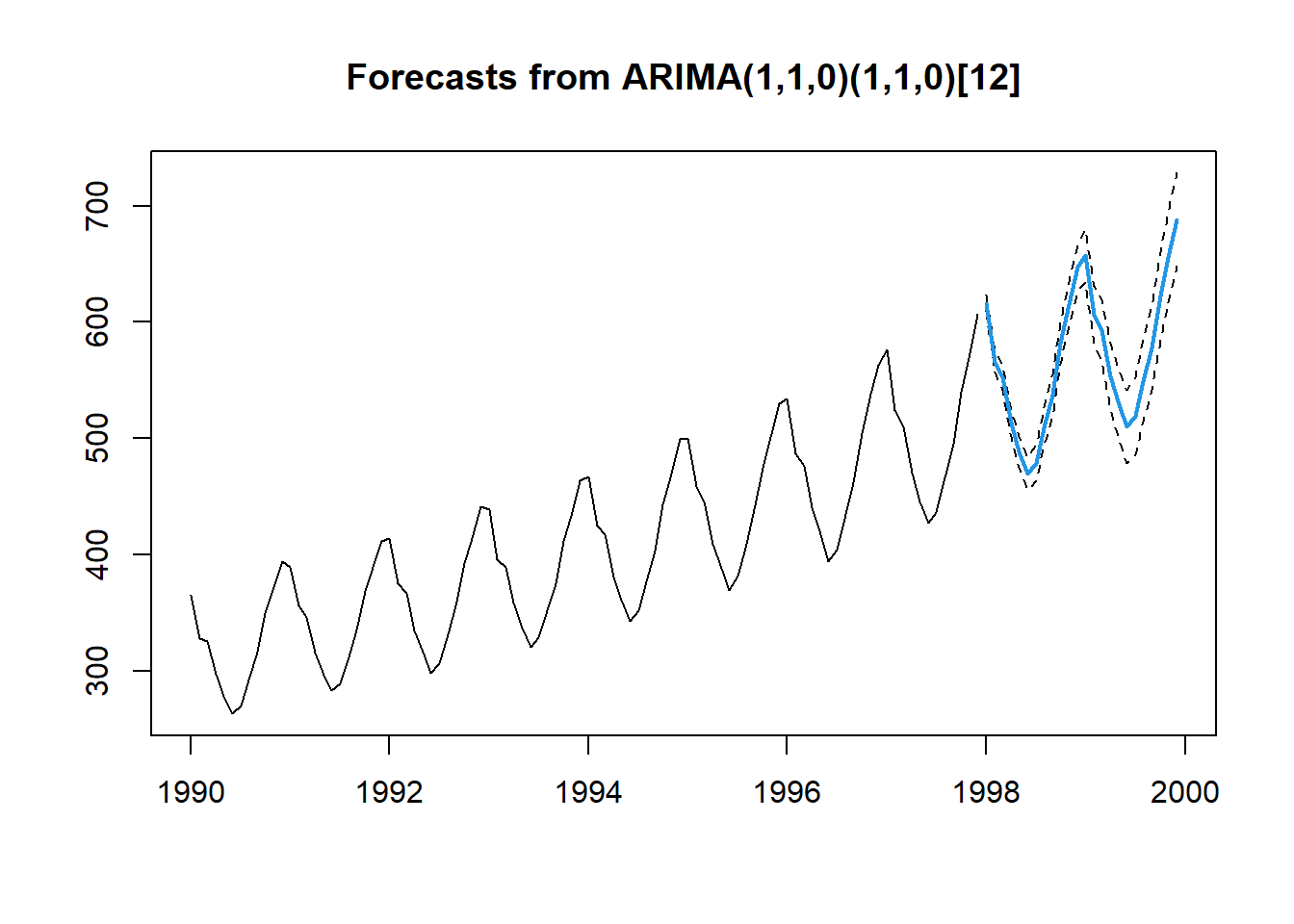
Bibliografía:
Cryer, J.D. y Chan, K.S. (2008). Time Series Analysis. With Applications in R. Springer.
Shumway, R.H. y Stoffer, D.S. (2006). Time Series Analysis and Its Applications. With R Examples. Springer.