6.4 Análisis de la varianza
6.4.1 ANOVA con un factor
Vamos a estudiar si hay diferencias en las medias de la variable satisfac (satisfacción global) entre los diferentes grupos definidos por nfidelid (nivel de compra), utilizando el procedimiento clásico de análisis de la varianza. Este procedimiento exige normalidad y homocedasticidad.
table(hatco$nfidelid)##
## bajo medio alto
## 3 64 33tapply(hatco$satisfac, hatco$nfidelid, mean, na.rm = TRUE)## bajo medio alto
## 3.533333 4.498437 5.443750La variable explicativa tiene que ser obligatoriamente de tipo factor. Por coherencia con la función (general) lm, la variación entre grupos está etiquetada nfidelid, y la variación dentro de los grupos como Residuals
anova(lm(satisfac~nfidelid, data = hatco))## Analysis of Variance Table
##
## Response: satisfac
## Df Sum Sq Mean Sq F value Pr(>F)
## nfidelid 2 23.832 11.9158 23.588 4.647e-09 ***
## Residuals 96 48.495 0.5052
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1Como alternativa, se puede utilizar la función aov
aov(satisfac~nfidelid, data = hatco)## Call:
## aov(formula = satisfac ~ nfidelid, data = hatco)
##
## Terms:
## nfidelid Residuals
## Sum of Squares 23.83161 48.49526
## Deg. of Freedom 2 96
##
## Residual standard error: 0.7107454
## Estimated effects may be unbalanced
## 1 observation deleted due to missingnesssummary(aov(satisfac~nfidelid, data = hatco))## Df Sum Sq Mean Sq F value Pr(>F)
## nfidelid 2 23.83 11.916 23.59 4.65e-09 ***
## Residuals 96 48.50 0.505
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
## 1 observation deleted due to missingnessComparaciones entre pares de variables
pairwise.t.test(hatco$satisfac, hatco$nfidelid)##
## Pairwise comparisons using t tests with pooled SD
##
## data: hatco$satisfac and hatco$nfidelid
##
## bajo medio
## medio 0.024 -
## alto 4.6e-05 5.5e-08
##
## P value adjustment method: holmRelajamos la hipótesis de varianzas iguales
oneway.test(satisfac~nfidelid, data = hatco)##
## One-way analysis of means (not assuming equal variances)
##
## data: satisfac and nfidelid
## F = 35.013, num df = 2.0000, denom df = 6.7661, p-value = 0.0002697Podemos utilizar el test de Bartlett para contrastar la igualdad de varianzas
bartlett.test(satisfac~nfidelid, data = hatco)##
## Bartlett test of homogeneity of variances
##
## data: satisfac by nfidelid
## Bartlett's K-squared = 1.4922, df = 2, p-value = 0.4742Representación gráfica
medias <- tapply(hatco$satisfac, hatco$nfidelid, mean, na.rm = TRUE)
desviaciones <- tapply(hatco$satisfac, hatco$nfidelid, sd, na.rm = TRUE)
n <- tapply(hatco$satisfac[!is.na(hatco$satisfac)], hatco$nfidelid[!is.na(hatco$satisfac)], length)
errores <- desviaciones/sqrt(n)
stripchart(hatco$satisfac~hatco$nfidelid, method='jitter', jit=0.01, pch=18, col='grey', vertical = TRUE)
arrows(1:3, medias+errores, 1:3, medias-errores, angle=90, code=3, lwd=2, col='orange')
points(1:3, medias, pch=4, lwd=2, cex=2, col='orange')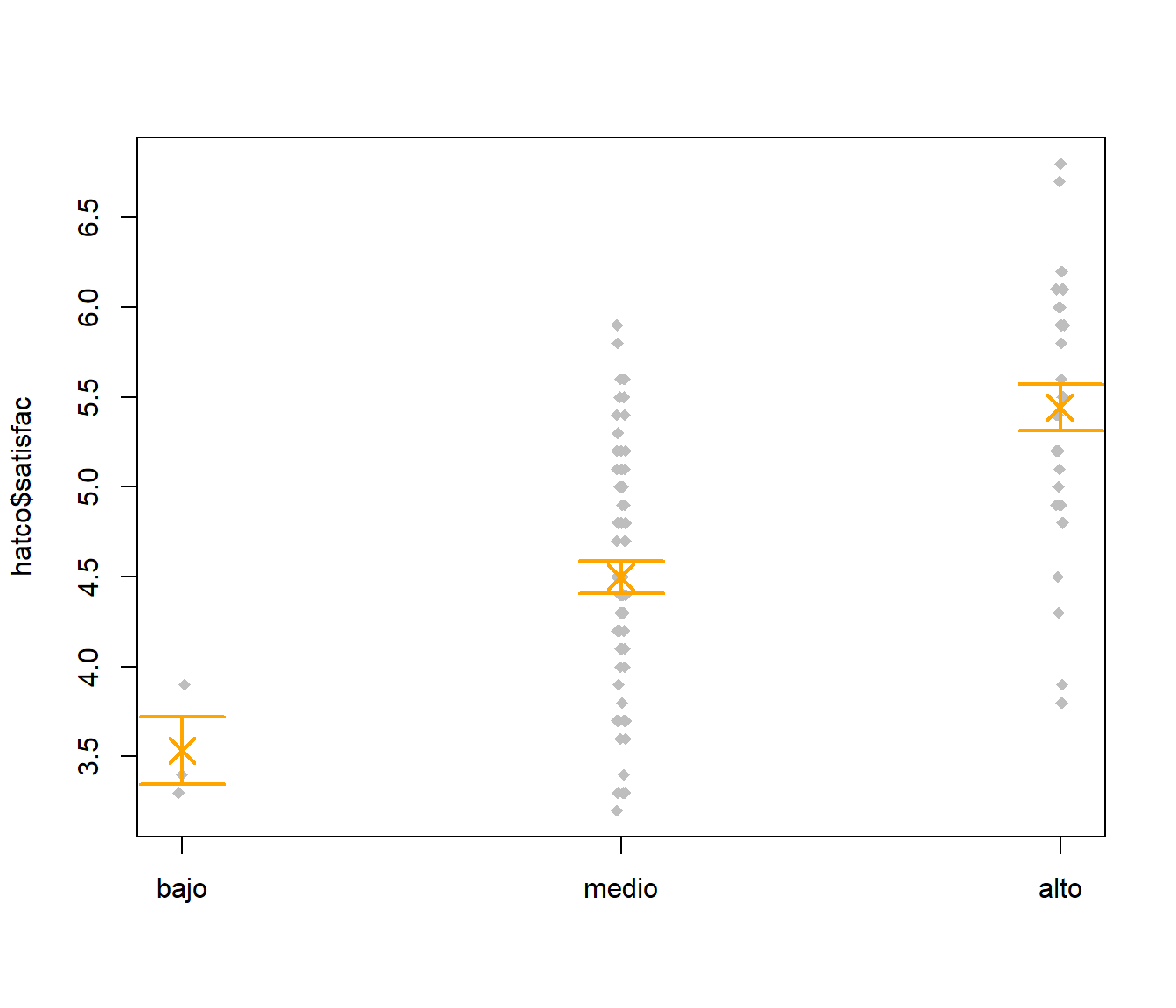
6.4.2 Test de Kruskal-Wallis
Alternativa no paramétrica al análisis de la varianza con un factor
kruskal.test(satisfac~nfidelid, data = hatco)##
## Kruskal-Wallis rank sum test
##
## data: satisfac by nfidelid
## Kruskal-Wallis chi-squared = 31.073, df = 2, p-value = 1.789e-07