8.5 Ejercicios
Ejercicio 8.1 (Bootstrap censurado por estratos) Analizar el conjunto de datos channing completo, teniendo en cuenta el sexo como estrato
(i.e. Surv(age, cens) ~ sex y strata = chan$sex)
# Datos
data(channing)
# Calcular edad (de partida o muerte) en años
channing$age <- (channing$entry + channing$time)/12
# Seleccionar variables
chan <-channing[c("age", "cens", "sex")]
# Estimación supervivencia
library(survival)
chan.F <- survfit(Surv(age, cens) ~ sex, data = chan)
chan.F## Call: survfit(formula = Surv(age, cens) ~ sex, data = chan)
##
## n events median 0.95LCL 0.95UCL
## sex=Female 365 130 88 86.7 89.5
## sex=Male 97 46 87 85.8 90.4plot(chan.F, lty = 1:2, xlim = c(60, 100))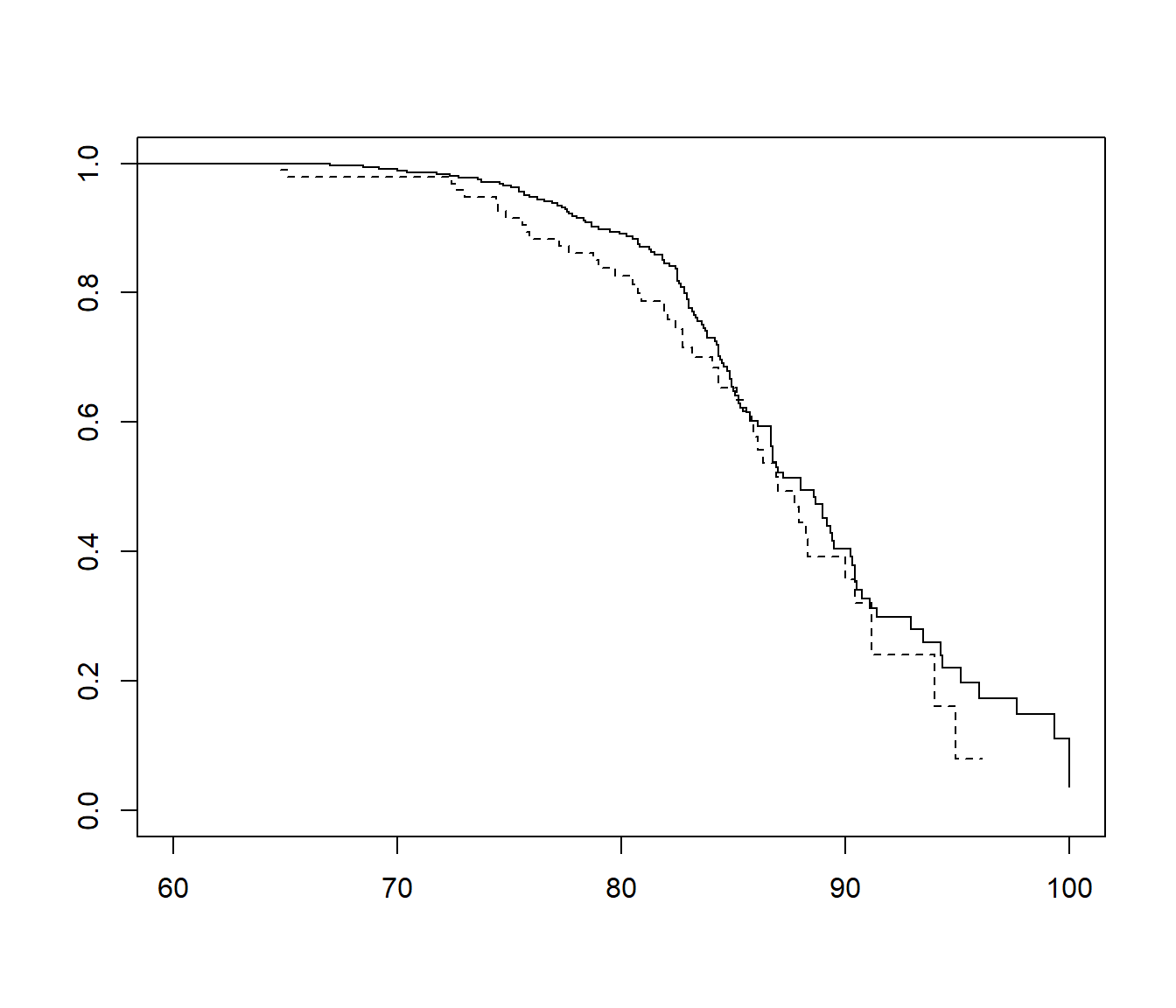
Figura 8.3: Estimaciones de la supervivencia.
res <- summary(chan.F)
# res
str(res)## List of 19
## $ n : int [1:2] 365 97
## $ time : num [1:146] 67 68.5 69.2 70 70.4 ...
## $ n.risk : num [1:146] 364 359 355 353 352 346 344 340 335 334 ...
## $ n.event : num [1:146] 1 1 1 1 1 1 1 1 1 1 ...
## $ n.censor : num [1:146] 2 3 3 1 0 6 0 3 4 0 ...
## $ surv : num [1:146] 0.997 0.994 0.992 0.989 0.986 ...
## $ std.err : num [1:146] 0.00274 0.0039 0.00479 0.00554 0.00619 ...
## $ cumhaz : num [1:146] 0.00275 0.00553 0.00835 0.01118 0.01402 ...
## $ std.chaz : num [1:146] 0.00275 0.00391 0.00482 0.00559 0.00627 ...
## $ strata : Factor w/ 2 levels "sex=Female","sex=Male": 1 1 1 1 1 1 1 1 1 1 ...
## $ type : chr "right"
## $ logse : logi TRUE
## $ conf.int : num 0.95
## $ conf.type : chr "log"
## $ lower : num [1:146] 0.992 0.987 0.982 0.978 0.974 ...
## $ upper : num [1:146] 1 1 1 1 0.998 ...
## $ call : language survfit(formula = Surv(age, cens) ~ sex, data = chan)
## $ table : num [1:2, 1:9] 365 97 365 97 365 ...
## ..- attr(*, "dimnames")=List of 2
## .. ..$ : chr [1:2] "sex=Female" "sex=Male"
## .. ..$ : chr [1:9] "records" "n.max" "n.start" "events" ...
## $ rmean.endtime: num [1:2] 101 101
## - attr(*, "class")= chr "summary.survfit"# Estimaciones de interés
res$table[, c("rmean", "median")]## rmean median
## sex=Female 88.46153 88
## sex=Male 86.89935 87as.numeric(res$table[, c("rmean", "median")])## [1] 88.46153 86.89935 88.00000 87.00000Ejercicio 8.2 (Bootstrap censurado con riesgo proporcional de Cox) Reproducir el ejemplo en Canty (2002, Rnews_2002-3) del modelo de riesgo proporcional de Cox (Cox, 1972):
# Datos
data(melanoma)
mel <- melanoma[melanoma$ulcer == 1, ]
mel$cens <- 1 * (mel$status == 1)
# Estimación supervivencia
library(survival)
# Modelo de riesgo proporcional de Cox
mel.cox <- coxph(Surv(time, cens) ~ thickness, data = mel)
mel.cox## Call:
## coxph(formula = Surv(time, cens) ~ thickness, data = mel)
##
## coef exp(coef) se(coef) z p
## thickness 0.09968 1.10481 0.04052 2.46 0.0139
##
## Likelihood ratio test=5 on 1 df, p=0.02541
## n= 90, number of events= 41# summary(mel.cox)
# Estadísticos de interés
mel.cox$coefficients## thickness
## 0.09967665