6.5 Evaluación de la precisión
Para evaluar la precisión de las predicciones podríamos utilizar el coeficiente de determinación ajustado:
summary(modelo)$adj.r.squared## [1] 0.7421059que estimaría la proporción de variabilidad explicada en una nueva muestra. Sin embargo, hay que tener en cuenta que su validez dependería de la de las hipótesis estructurales (especialmente de la linealidad, homocedasticidad e independencia), ya que se obtiene a partir de estimaciones de las varianzas residual y total:
\[R_{ajus}^{2} = 1 - \frac{\hat{S}_{R}^{2}}{\hat{S}_{Y}^{2}} = 1 - \left( \frac{n-1}{n-p-1} \right) (1-R^{2})\]
siendo \(\hat{S}_{R}^{2}=\sum_{i=1}^{n}(y_{i}-\hat{y}_{i})^{2}/(n - p - 1)\). Algo similar ocurriría con otras medidas de bondad de ajuste, como por ejemplo BIC o AIC.
Alternativamente, por si no es razonable asumir estas hipótesis, se pueden emplear el procedimiento tradicional en AE (o alguno de los otros descritos en la Sección 1.3).
Podemos evaluar el modelo ajustado en el conjunto de datos de test y comparar las predicciones frente a los valores reales (ver Figura 6.10):
obs <- test$fidelida
pred <- predict(modelo, newdata = test)
plot(pred, obs, xlab = "Predicción", ylab = "Observado")
abline(a = 0, b = 1)
res <- lm(obs ~ pred) # summary(res)
abline(res, lty = 2)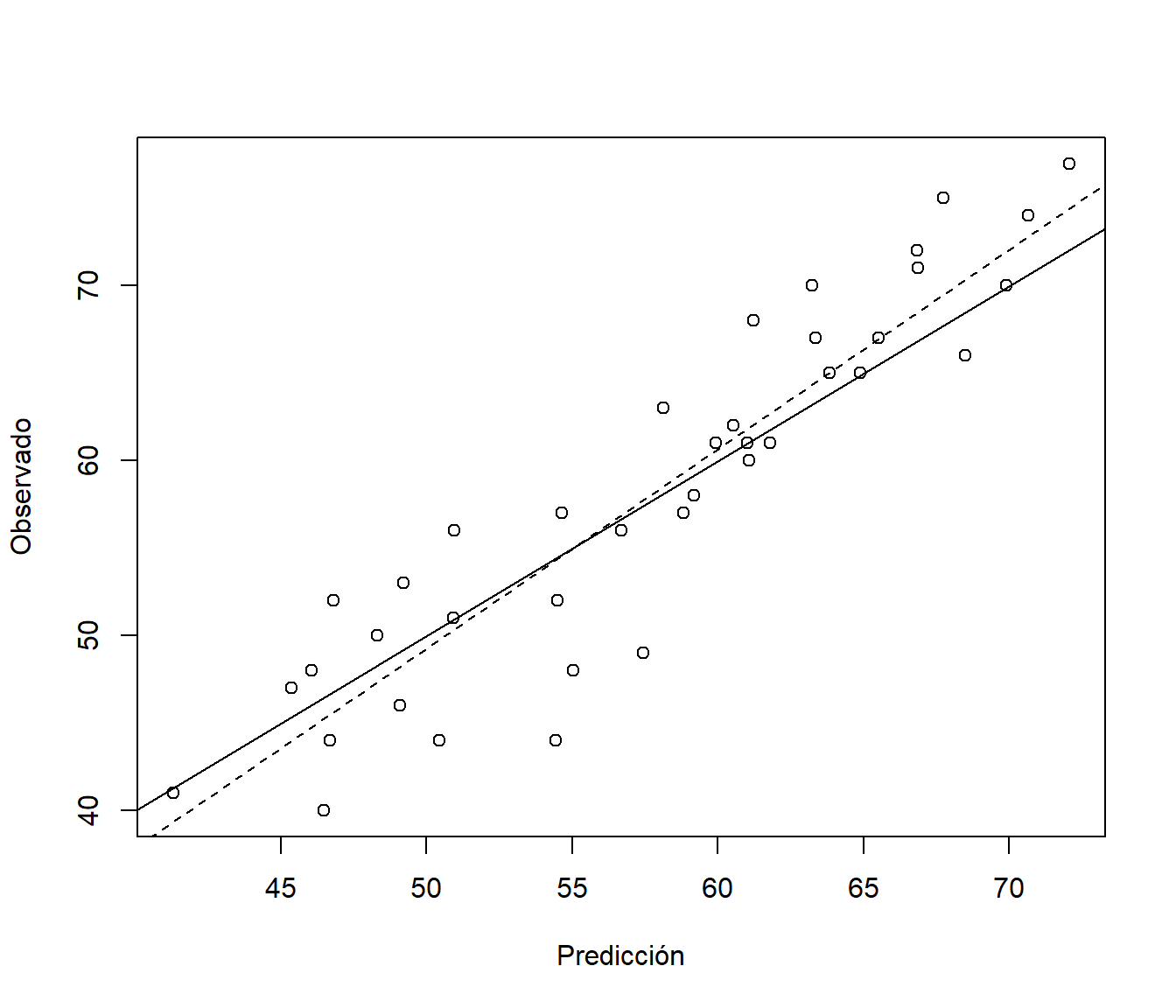
Figura 6.10: Gráfico de dispersión de observaciones frente a predicciones, del ajuste lineal en la muestra de test.
También podemos obtener medidas de error, por ejemplo empleando la función accuracy() de la Sección 1.3.4:
accuracy(pred, obs)## me rmse mae mpe mape r.squared
## 0.4032996 4.1995208 3.3013714 -0.1410512 5.9553699 0.8271449