1.6 Introducción al paquete caret
Como ya se comentó en la Sección 1.2.2, el paquete caret (Classification And REgression Training, Kuhn, 2008; ver también Kuhn, 2019) proporciona una interfaz unificada que simplifica el proceso de modelado empleando la mayoría de los métodos de AE implementados en R (actualmente admite 239 métodos, listados en el Capítulo 6 de Kuhn, 2019). Además de proporcionar rutinas para los principales pasos del proceso, incluye también numerosas funciones auxiliares que permiten implementar nuevos procedimientos. Este paquete ha dejado de desarrollarse de forma activa y se espera que en un futuro próximo sea sustituido por el paquete tidymodels (ver Kuhn y Silge, 2022), aunque hemos optado por utilizarlo en este libro porque consideramos que esta alternativa aún se encuentra en fase de desarrollo y además requiere de mayor tiempo de aprendizaje.
En esta sección se describirán de forma esquemática las principales herramientas disponibles en este paquete, para más detalles se recomienda consultar el manual (Kuhn, 2019). También está disponible una pequeña introducción en la vignette del paquete: A Short Introduction to the caret Package19.
La función principal es train(), que incluye un parámetro method que permite establecer el modelo mediante una cadena de texto. Podemos obtener información sobre los modelos disponibles con las funciones getModelInfo() y modelLookup() (puede haber varias implementaciones del mismo método con distintas configuraciones de hiperparámetros; también se pueden definir nuevos modelos, ver el Capítulo 13 del manual).
library(caret)
str(names(getModelInfo())) # Listado de los métodos disponibles## chr [1:239] "ada" "AdaBag" "AdaBoost.M1" "adaboost" ...# names(getModelInfo("knn")) # Encuentra 2 métodos
modelLookup("knn") # Información sobre hiperparámetros## model parameter label forReg forClass probModel
## 1 knn k #Neighbors TRUE TRUE TRUEEn la siguiente tabla se muestran los métodos actualmente disponibles:
Figura 1.17: Listado de los métodos disponiles en caret::train().
El paquete caret permite, entre otras cosas:
Partición de los datos:
createDataPartition(y, p = 0.5, list = TRUE, ...): crea particiones balanceadas de los datos.En el caso de que la respuesta
ysea categórica, realiza el muestreo en cada clase. Para respuestas numéricas emplea cuantiles (definidos por el argumentogroups = min(5, length(y))).p: proporción de datos en la muestra de entrenamiento.list: lógico; determina si el resultado es una lista con las muestras o un vector (o matriz) de índices.
Funciones auxiliares:
createFolds(),createMultiFolds(),groupKFold(),createResample(),createTimeSlices()
Análisis descriptivo:
featurePlot()Preprocesado de los datos:
La función principal es
preProcess(x, method = c("center", "scale"), ...), aunque se puede integrar en el entrenamiento (funcióntrain()). Estimará los parámetros de las transformaciones con la muestra de entrenamiento y permitirá aplicarlas posteriormente de forma automática al hacer nuevas predicciones (por ejemplo, en la muestra de test).El parámetro
methodpermite establecer una lista de procesados:Imputación:
"knnImpute","bagImpute"o"medianImpute".Creación y transformación de variables explicativas:
"center","scale","range","BoxCox","YeoJohnson","expoTrans","spatialSign".Selección de predictores y extracción de componentes:
"corr","nzv","zv","conditionalX","pca","ica".
Dispone de múltiples funciones auxiliares, como
dummyVars()orfe()(recursive feature elimination).
Entrenamiento y selección de los hiperparámetros del modelo:
La función principal es
train(formula, data, method = "rf", trControl = trainControl(), tuneGrid = NULL, tuneLength = 3, metric, ...)trControl: permite establecer el método de remuestreo para la evaluación de los hiperparámetros y el método para seleccionar el óptimo, incluyendo las medidas de precisión. Por ejemplo,trControl = trainControl(method = "cv", number = 10, selectionFunction = "oneSE").Los métodos disponibles son:
"boot","boot632","optimism_boot","boot_all","cv","repeatedcv","LOOCV","LGOCV","timeslice","adaptive_cv","adaptive_boot"o"adaptive_LGOCV".tuneLengthytuneGrid: permite establecer cuántos hiperparámetros serán evaluados (por defecto 3) o una rejilla con las combinaciones de hiperparámetros.metric: determina el criterio para la selección de hiperparámetros. Por defecto,metric = "RMSE"en regresión ometric = "Accuracy"en clasificación. Sin modificar otras opciones20 también se podría establecermetric = "Rsquared"para regresión ymetric = "Kappa"en clasificación....permite establecer opciones específicas de los métodos.
También admite matrices
x,yen lugar de fórmulas (o recetas:recipe()).Si se imputan datos en el preprocesado será necesario establecer
na.action = na.pass.
Predicción: Una de las ventajas es que incorpora un único método
predict()para objetos de tipotraincon dos únicas opciones21type = c("raw", "prob"), la primera para obtener predicciones de la respuesta y la segunda para obtener estimaciones de las probabilidades (en los métodos de clasificación que lo admitan).Además, si se incluyó un preprocesado en el entrenamiento, se emplearán las mismas transformaciones en un nuevo conjunto de datos
newdata.Evaluación de los modelos:
postResample(pred, obs): regresión.confusionMatrix(pred, obs, ...): clasificación.
Análisis de la importancia de los predictores:
varImp(): interfaz a las medidas específicas de los métodos de aprendizaje supervisado (Sección 15.1 del manual) o medidas genéricas (Sección 15.2).
Como ejemplo consideraremos el problema de regresión anterior, empleando KNN en caret:
data(Boston, package = "MASS")
library(caret)En primer lugar, particionamos los datos:
set.seed(1)
itrain <- createDataPartition(Boston$medv, p = 0.8, list = FALSE)
train <- Boston[itrain, ]
test <- Boston[-itrain, ]Realizamos el entrenamiento, incluyendo un preprocesado de los datos (se almacenan las transformaciones para volver a aplicarlas en la predicción con nuevos datos) y empleando validación cruzada con 10 grupos para la selección de hiperparámetros (ver Figura 1.18). Además, en lugar de utilizar las opciones por defecto, establecemos la rejilla de búsqueda del hiperparámetro:
set.seed(1)
knn <- train(medv ~ ., data = train, method = "knn",
preProc = c("center", "scale"), tuneGrid = data.frame(k = 1:10),
trControl = trainControl(method = "cv", number = 10))
knn## k-Nearest Neighbors
##
## 407 samples
## 13 predictor
##
## Pre-processing: centered (13), scaled (13)
## Resampling: Cross-Validated (10 fold)
## Summary of sample sizes: 367, 366, 367, 366, 365, 367, ...
## Resampling results across tuning parameters:
##
## k RMSE Rsquared MAE
## 1 4.6419 0.74938 3.0769
## 2 4.1140 0.79547 2.7622
## 3 3.9530 0.81300 2.7041
## 4 4.2852 0.78083 2.8905
## 5 4.6161 0.75187 3.0647
## 6 4.7543 0.73863 3.1622
## 7 4.7346 0.74041 3.1515
## 8 4.6563 0.75083 3.1337
## 9 4.6775 0.75082 3.1567
## 10 4.6917 0.74731 3.2076
##
## RMSE was used to select the optimal model using the smallest value.
## The final value used for the model was k = 3.ggplot(knn, highlight = TRUE) # Alternativamente: plot(knn)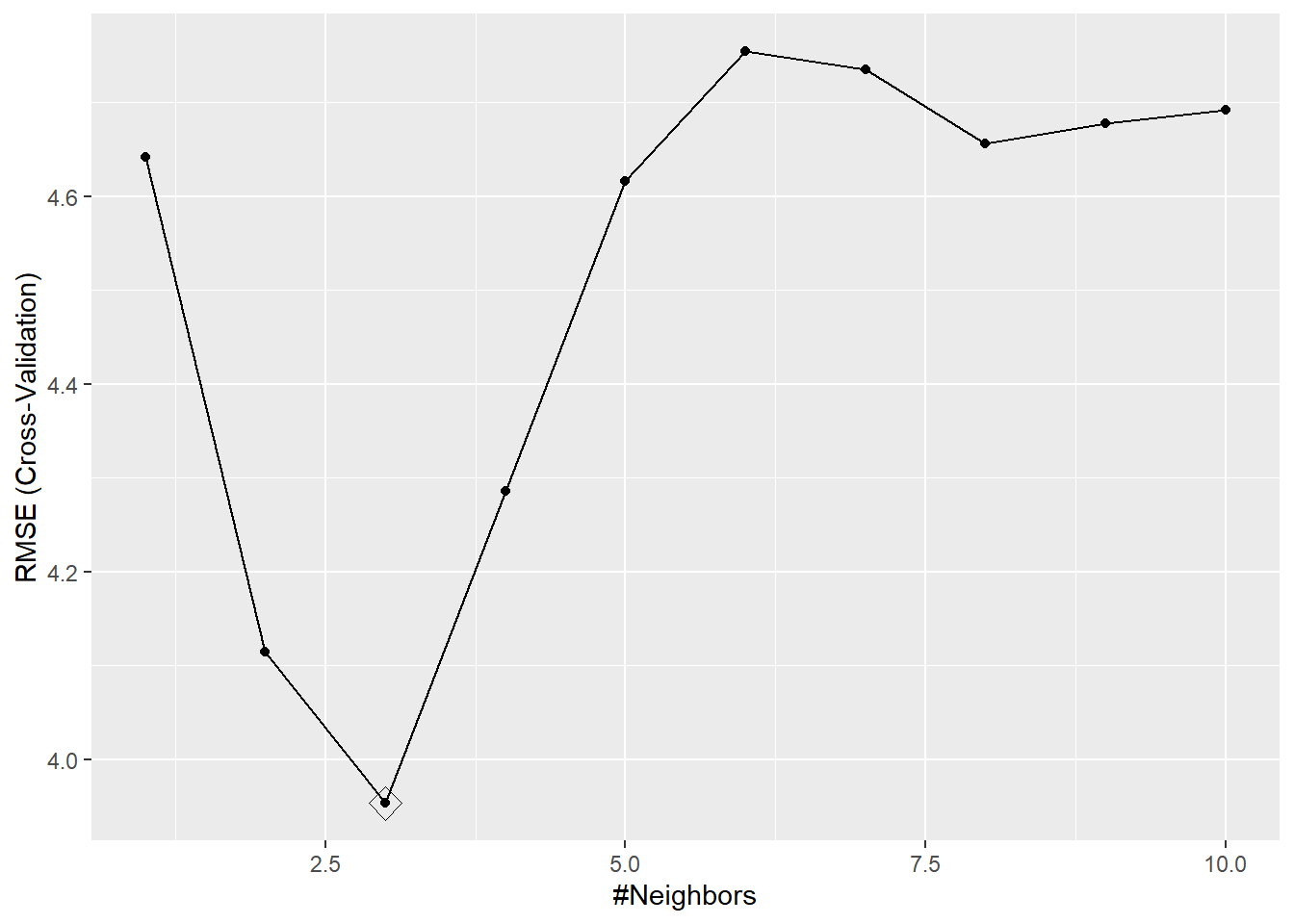
Figura 1.18: Raíz del error cuadrático medio de validación cruzada dependiendo del valor del hiperparámetro, resaltando el valor óptimo.
Los valores seleccionados de los hiperparámetros se devuelven en la componente $bestTune:
knn$bestTune## k
## 3 3y en la componente $finalModel el modelo final ajustado (en el formato del paquete que se empleó internamente para el ajuste):
knn$finalModel## 3-nearest neighbor regression modelObtenemos medidas de la importancia de las variables (interpretación del modelo):
varImp(knn)## loess r-squared variable importance
##
## Overall
## lstat 100.0
## rm 88.3
## indus 36.3
## ptratio 33.3
## tax 30.6
## crim 28.3
## nox 23.4
## black 21.3
## age 20.5
## rad 17.2
## zn 15.1
## dis 14.4
## chas 0.0y, finalmente, evaluamos la capacidad predictiva del modelo obtenido empleando la muestra de test:
postResample(predict(knn, newdata = test), test$medv)## RMSE Rsquared MAE
## 4.96097 0.73395 2.72424Bibliografía
Accesible con el comando
vignette("caret"). También puede resultar de interés la “chuleta” https://github.com/rstudio/cheatsheets/blob/main/caret.pdf.↩︎Para emplear medidas adicionales habría que definir la función que las calcule mediante el argumento
summaryFunctiondetrainControl(), como se indica en el Ejercicio 5.3.↩︎En lugar de la variedad de opciones que emplean los distintos paquetes (p. ej.:
type = "response","class","posterior","probability"…).↩︎