8.3 Implementación en R
Hay numerosos paquetes que implementan redes neuronales, aunque por simplicidad utilizaremos el paquete nnet (Venables y Ripley, 2002) que implementa redes neuronales feedfordward con una única capa oculta y está incluido en el paquete base de R.
Para el caso de redes más complejas se puede utilizar, por ejemplo, el paquete neuralnet (Fritsch et al., 2019), pero en el caso de grandes volúmenes de datos o aprendizaje profundo la recomendación sería emplear paquetes computacionalmente más eficientes (con computación en paralelo empleando CPUs o GPUs) como keras, h2o o sparlyr, entre otros.
La función principal nnet() se suele emplear con los siguientes argumentos:
nnet(formula, data, size, Wts, linout = FALSE, skip = FALSE,
rang = 0.7, decay = 0, maxit = 100, ...)formulaydata(opcional): permiten especificar la respuesta y las variables predictoras de la forma habitual (p. g.ej.respuesta ~ .; también implementa una interfaz con matricesxey). Admite respuestas multidimensionales (ajustará un modelo para cada componente) y categóricas (las convierte en multivariantes si tienen más de dos categorías y emplea softmax en los nodos finales). Teniendo en cuenta que por defecto los pesos iniciales se asignan al azar (Wts <- runif(nwts, -rang, rang)), la recomendación sería reescalar los predictores en el intervalo \([0, 1]\), sobre todo si se emplea regularización (decay > 0).size: número de nodos en la capa oculta.linout: permite seleccionar la identidad como función de activación en los nodos finales; por defectoFALSEy empleará la función logística o softmax en el caso de factores con múltiples niveles (si se emplea la interfaz de fórmula, con matrices habrá que establecersoftmax = TRUE).skip: permite añadir pesos adicionales entre la capa de entrada y la de salida (saltándose la capa oculta); por defectoFALSE.decay: parámetro \(\lambda\) de regularización de los pesos (weight decay); por defecto 0. Para emplear este parámetro los predictores deberían estar en la misma escala.maxit: número máximo de iteraciones; por defecto 100.
Como ejemplo consideraremos el conjunto de datos earth::Ozone1 empleado en el capítulo anterior:
data(ozone1, package = "earth")
df <- ozone1
set.seed(1)
nobs <- nrow(df)
itrain <- sample(nobs, 0.8 * nobs)
train <- df[itrain, ]
test <- df[-itrain, ]En este caso, emplearemos el método "nnet" de caret para preprocesar los datos y seleccionar el número de nodos en la capa oculta y el parámetro de regularización.
Como caret emplea las opciones por defecto de nnet() (diseñadas para clasificación),
estableceremos linout = TRUE71 y aumentaremos el número de iteraciones (aunque seguramente siga siendo demasiado pequeño).
library(caret)
# getModelInfo("nnet") # Encuentra 10 métodos con "nnet"
modelLookup("nnet")## model parameter label forReg forClass probModel
## 1 nnet size #Hidden Units TRUE TRUE TRUE
## 2 nnet decay Weight Decay TRUE TRUE TRUEtuneGrid <- expand.grid(size = 2*1:5, decay = c(0, 0.001, 0.01))
set.seed(1)
caret.nnet <- train(O3 ~ ., data = train, method = "nnet",
preProc = c("range"), # Reescalado en [0,1]
tuneGrid = tuneGrid,
trControl = trainControl(method = "cv", number = 10),
linout = TRUE, maxit = 200, trace = FALSE)En este caso se seleccionaron 4 nodos en la capa oculta y un valor de 0.01 para el parámetro de regularización (ver Figura 8.2).
ggplot(caret.nnet, highlight = TRUE)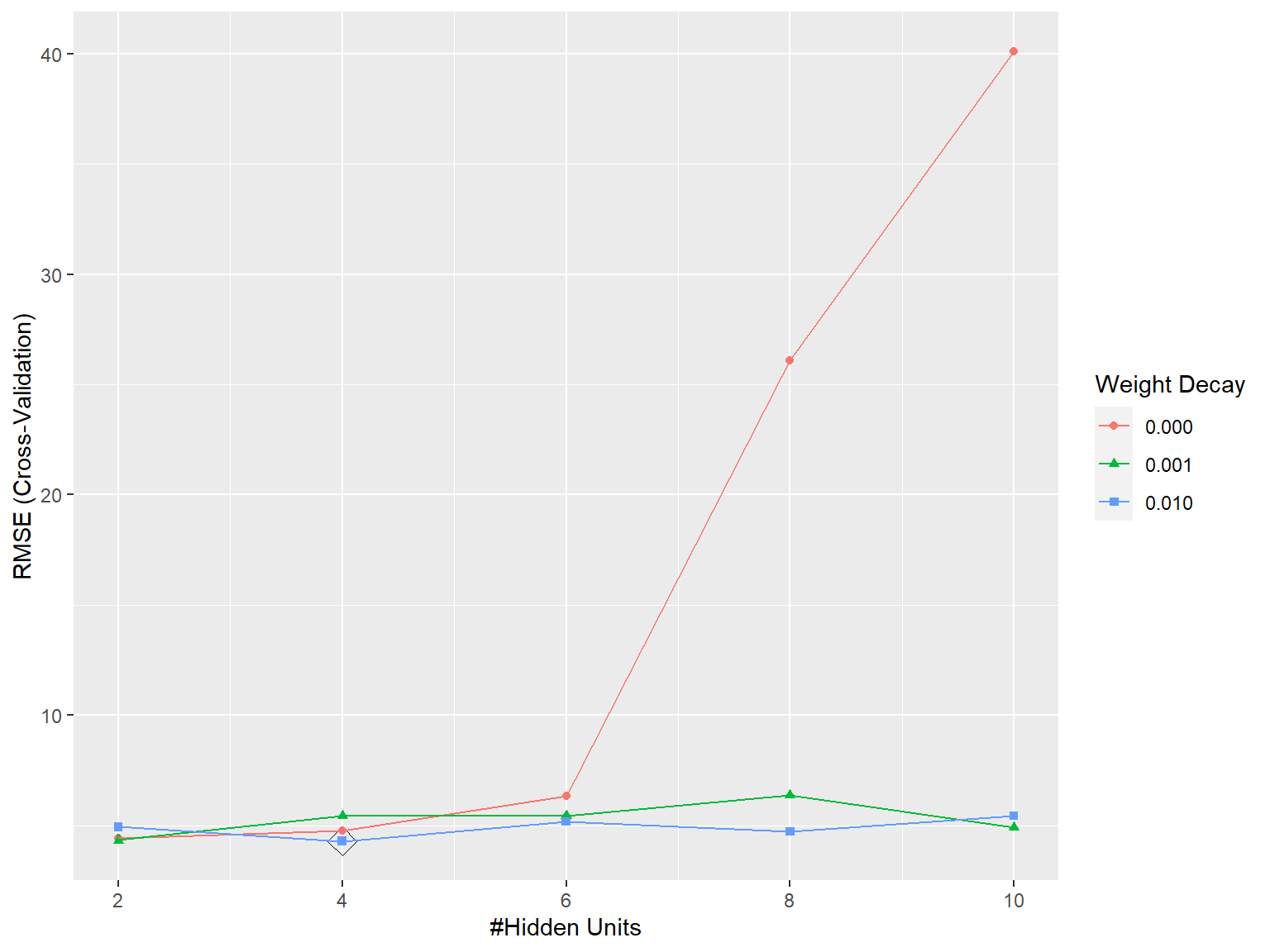
Figura 8.2: Selección de los hiperparámetros asociados a una red neuronal (el número de nodos y el parámetro de regularización) mediante un criterio de error RMSE calculado por validación cruzada.
A continuación, analizamos el modelo resultante con el método summary():
summary(caret.nnet$finalModel)## a 9-4-1 network with 45 weights
## options were - linear output units decay=0.01
## b->h1 i1->h1 i2->h1 i3->h1 i4->h1 i5->h1 i6->h1 i7->h1 i8->h1 i9->h1
## -8.66 3.74 -5.50 -18.11 -12.83 6.49 14.39 -4.53 14.48 -1.96
## b->h2 i1->h2 i2->h2 i3->h2 i4->h2 i5->h2 i6->h2 i7->h2 i8->h2 i9->h2
## -2.98 1.78 0.00 1.58 1.96 -0.60 0.63 2.46 2.36 -19.69
## b->h3 i1->h3 i2->h3 i3->h3 i4->h3 i5->h3 i6->h3 i7->h3 i8->h3 i9->h3
## 25.23 -50.14 9.74 -3.66 -5.61 4.21 -11.17 39.34 -20.18 0.37
## b->h4 i1->h4 i2->h4 i3->h4 i4->h4 i5->h4 i6->h4 i7->h4 i8->h4 i9->h4
## -3.90 4.94 -1.08 1.50 1.52 -0.54 0.14 -1.27 0.98 -1.54
## b->o h1->o h2->o h3->o h4->o
## -5.32 4.19 -14.03 7.50 38.75que muestra los valores de los pesos (45 en total).
Aunque suele ser preferible representarlo gráficamente, empleando el paquete NeuralNetTools (Beck, 2018), ver Figura 8.3:
library(NeuralNetTools)
plotnet(caret.nnet$finalModel)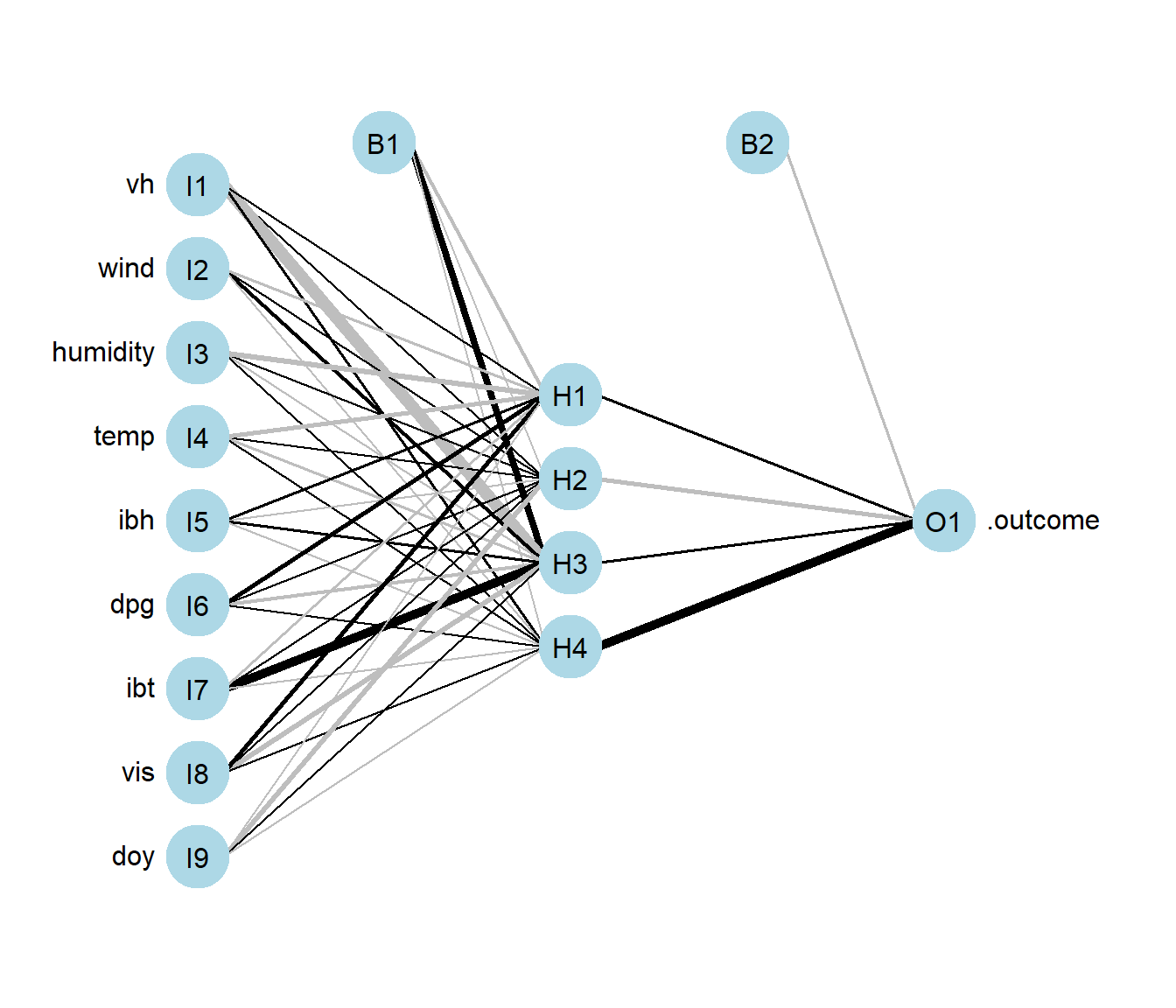
Figura 8.3: Representación de la red neuronal ajustada (generada con el paquete NeuralNetTools; con colores y grosores según el signo y magnitud de los pesos).
Por último, evaluamos las predicciones en la muestra de test:
pred <- predict(caret.nnet, newdata = test)
obs <- test$O3
library(mpae)
accuracy(pred, obs)## me rmse mae mpe mape r.squared
## 0.33213 3.02422 2.44670 -7.40960 32.80001 0.85865y las representamos gráficamente (ver Figura 8.4):
pred.plot(pred, obs, xlab = "Predicción", ylab = "Observado")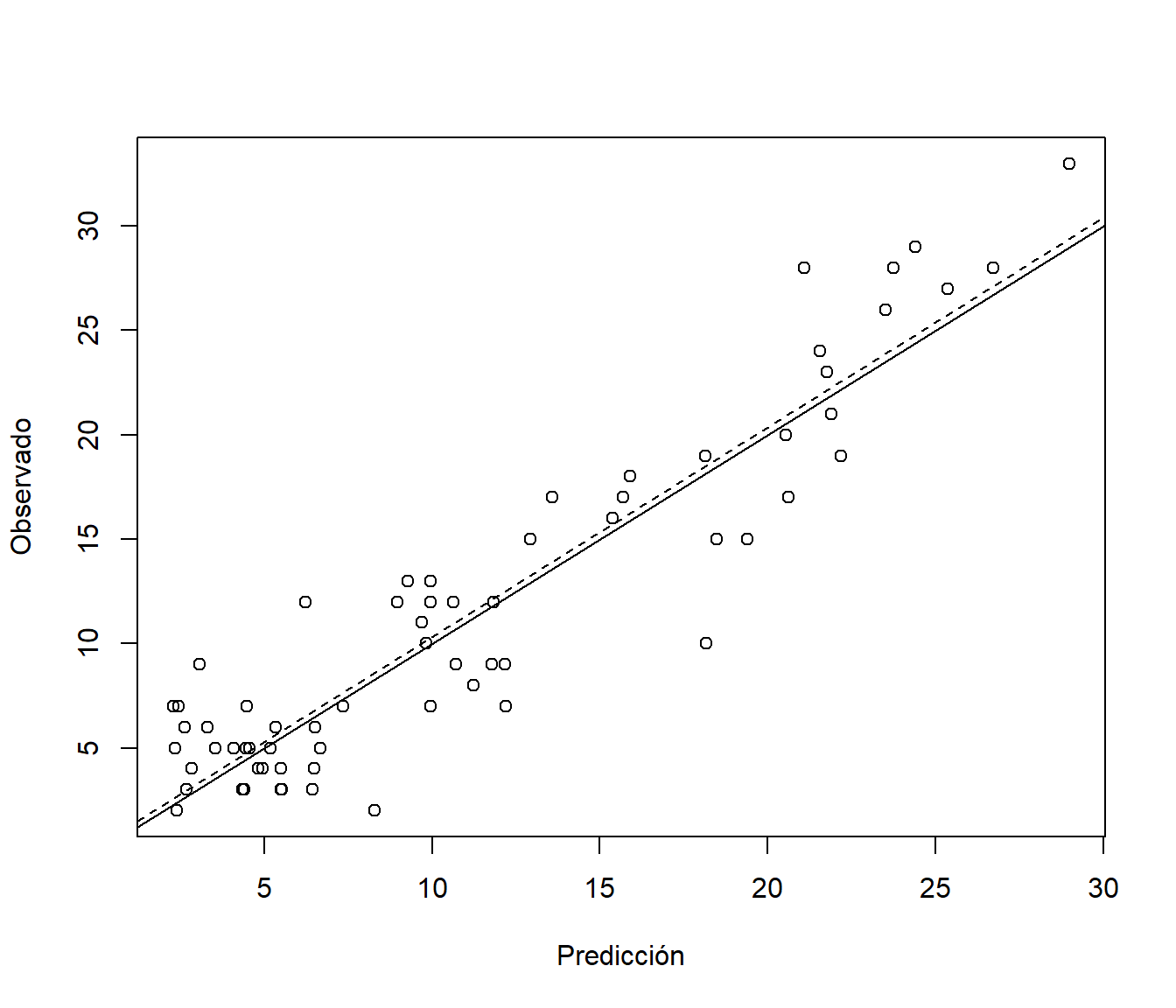
Figura 8.4: Observaciones frente a predicciones (en la muestra de test) con la red neuronal ajustada.
Ejercicio 8.1 Continuando con el conjunto de datos mpae::bodyfat empleado en capítulos anteriores, particiona los datos y ajusta una red neuronal para predecir el porcentaje de grasa corporal (bodyfat), mediante el método nnet de caret:
Fija el parámetro de penalización
decay = 0.001y selecciona el número de nodos en la capa ocultasize = 2:5mediante validación cruzada con 10 grupos, considerando un máximo de 125 iteraciones en el algoritmo de aprendizaje.Representa gráficamente la red obtenida e indica el número de parámetros del modelo.
Evalúa su precisión en la muestra de test y compara los resultados con los obtenidos en la Sección 2.1.4 o en ejercicios anteriores.
Ejercicio 8.2 Continuando con el Ejercicio 7.2, que empleaba el conjunto de datos iris como ejemplo de un problema de clasificación multiclase, utiliza el método nnet de caret para clasificar la especie de lirio (Species) a partir de las dimensiones de los sépalos y pétalos de sus flores.
Considera el 80 % de las observaciones como muestra de aprendizaje y el 20 % restante como muestra de test.
Fija el parámetro de regularización de los pesos decay = 0.001 y selecciona el número de nodos en la capa oculta size = seq(2, 10, by = 2) de forma que se minimice el error de clasificación de validación cruzada con 10 grupos.
Representa gráficamente la red obtenida e indica el número de parámetros del modelo.
Finalmente, evalúa la precisión de las predicciones en la muestra test.
Bibliografía
La alternativa sería transformar la respuesta a rango 1.↩︎